![]()
COMPUTACION BIOLOGICA o el por qué una BACTERIA y un SUPER COMPUTADOR son tan parecidos
por Mario Aguilar Herrador
Alan Turing, matemático y criptógrafo británico, el cual es considerado uno de los padres de la informática, ya flirteaba en los años 50 con la posibilidad de utilizar ordenadores para entender mejor los procesos biológicos complejos. En 1959, Richard Feynman dio una visionaria charla describiendo la posibilidad de construir ordenadores que era “submicroscópicos”.
Desde el desarrollo de modelos matemáticos sobre la morfogénesis (proceso por el cual un organismo adquiere una forma determinada); hasta la inteligencia artificial, la cual pretende imitar las capacidades cognitivas de la mente humana, nacen dos ramas del conocimiento que, a pesar de sonar igual, no lo son tanto: la biología computacional y la computación biológica.
La biología computacional es una ciencia que recurre a herramientas informáticas para ayudarnos a entender los sistemas biológicos, mientras que la computación biológica estudia cómo utilizar elementos de naturaleza biológica para procesar y almacenar la información, así como inspirar nuevos algoritmos replicando los mecanismos de la evolución biológica.
Si pensamos en términos de un ordenador, el término que nos atañe utiliza como hardware las proteínas o el ADN para llevar a cabo cálculos, escribir y leer la información. Si pensamos en el software, destacamos tres principales fuentes de inspiración:
– La población. Tanto una bacteria como una persona no se comporta igual si está sola o aislada que si está en un grupo reducido o entre una multitud. La computación biológica se inspira en este concepto para entender y predecir el comportamiento de las poblaciones formadas por individuos del mismo tipo que tienen la capacidad de interactuar.
– El conexionismo. Propone que los procesos mentales que explican nuestras capacidades cognitivas, así como nuestro comportamiento, se pueden describir por unidades sencillas iguales entre sí. Biológicamente, estas unidades son las neuronas, que establecen sinapsis entre sí, enfoque que ha servido para el desarrollo de redes neuronales artificiales.
– La emergencia. O, dicho de otra forma, un organismo es más que la suma de sus partes. Las neuronas, por sí solas, no tienen consciencia, pero es el conjunto de todas las reacciones desencadenadas por la comunicación sináptica el que hace que la consciencia emerja de nuestra mente.
Aunque aún estamos lejos de que los ordenadores resuelvan problemas biológicos por nosotros, la nanobiotecnología ha impulsado esto fuertemente. Esta ciencia es la que permite ensamblar proteínas con estructuras funcionales más complejas. De esta forma, se han puesto en marcha los primeros ordenadores biológicos que tienen la capacidad de realizar cálculos manipulando el ARN (el primo hermano del ADN) de una bacteria. Expuesto así, lejos queda el silicio y el coltán de nuestros microprocesadores y móviles, pero no es ningún tipo de prestidigitación. Esta tecnología consiste en diseñar un circuito biológico manipulando moléculas de ADN como si fuera un circuito digital, implementando las mismas operaciones lógicas que llevan a cabo los procesadores de silicio convencionales.
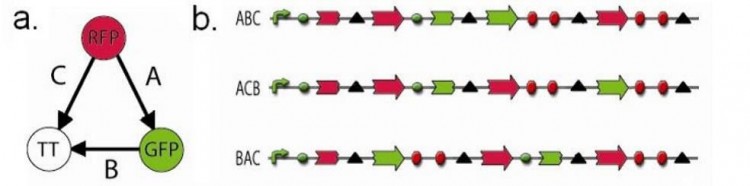
Figura 1. Ejemplo de un circuito genético. En este caso, la resolución permite a la bacteria evaluar todas las posibilidades del grafo dirigido para encontrar un camino Hamiltoniano (a). Los constructos representan respuestas posibles al problema planteado, siendo el constructo ABC la única solución posible al obtener las secuencias de los genes RFP y GFP en orden y orientación correcta (b).
Una vez este circuito biológico está preparado, se introduce en forma de plásmido en una bacteria Escherichia coli mediante un proceso de transformación. Esta bacteria, es la bacteria modelo por antonomasia, ya que ha sido ampliamente caracterizada y estudiada, y las que se utilizan en laboratorios son idénticas a las que viven n nuestro intestino. El ADN donde está nuestro circuito biológico atraviesa la pared bacteriana mediante el proceso de transformación y llega al interior, donde se lee y se traduce a una molécula de ARN mensajero (ARNm) las cuales serán traducidas de nuevo por un orgánulo celular denominado ribosoma, fabricándose así la proteína de interés. La miga del asunto es que las proteínas se sintetizan en función de una entrada concreta (entrada estándar) y la proteína, será la salida (salida estándar), simulando el comportamiento de un transistor, y formando una puerta lógica entre el ARNm y el ribosoma, el cual es el dispositivo electrónico mínimo de nuestros microprocesadores que es capaz de llevar una operación o calculo.
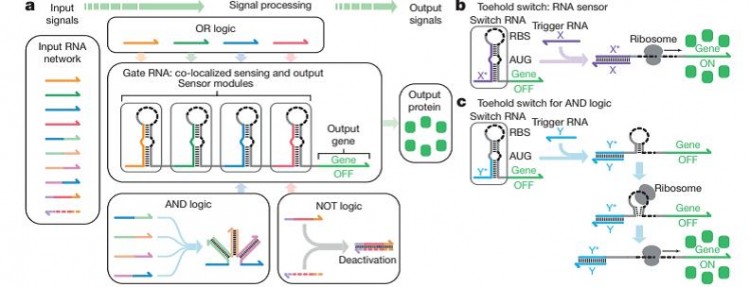
Alexander A. Green y su grupo consiguió utilizar esta tecnología para poner a punto un circuito biológico constituido por cinco puertas lógicas OR, otras cinco AND y dos NOT. No es el mejor procesador del mercado (ni está cerca) pero por algún sitio se empieza para conseguir un ordenador biológico capaz de llevar a cabo procesos complejos.
La computación biológica, en el ámbito del hardware es una promesa a largo plazo, pero en el caso del software, la cosa es muy distinta. Las herramientas más interesantes proponen sistemas basados en el aprendizaje de poblaciones o en algoritmos genéticos. Sin embargo, mucho más notable es el caso de Tierra, una simulación desarrollada en 1991 por el biólogo Thomas Ray, en la que un ordenador simulado ejecuta una serie de programas informáticos que se replican, están sujetos a mutaciones y compiten entre sí por los recursos del ordenador. Esto es similar a la lectura y traducción de un genoma bacteriano a proteína. El resultado es un proceso evolutivo totalmente controlable y fácilmente analizable. El propio Ray despertó cierta polémica al armar que los programas informáticos que evolucionaban en su simulación no imitaban procesos biológicos, sino que directamente podían considerarse como seres vivos a todos los efectos. A pesar de esto, los algoritmos genéticos no son la mejor opción para resolver cualquier problema. Su estrategia provoca que en ocasiones no nos entreguen la solución óptima, que nos devuelvan una solución que no es satisfactoria, o, incluso, que su coste en recursos y tiempo de computación sea inasumible.
Figura 2. Esquema publicado por Alexander Green y sus colaboradores. En él, se muestra un sistema de computación in vivo usando dispositivos “ribocomputadores”. Las moléculas de ARN son la entrada y la proteína es la señal, es decir, en función del tipo de molécula de ARN que se introduzca, la lógica hará que se formen estructuras determinadas que harán que se expresen proteínas diferentes. Por ejemplo, la puerta NOT, implica que, si se tiene el ARN en un sentido y su complementario, se desactivará la expresión del gen de la proteína.
Como respuesta a esto, tenemos las redes neuronales artificiales, que son una de las herramientas con mayor potencial que se utilizan para implementar sistemas de aprendizaje automático. Su funcionamiento se parece al de nuestro cerebro y la complejidad reside esencialmente en las operaciones que puede llevar a cabo cada neurona artificial a partir de los datos de entrada que recibe, como están conectadas las neuronas y como interaccionan entre ellas. Lo más asombroso es que no necesitan ser programadas de forma explícita, es decir, que son capaces de aprender por si misma a partir de un conjunto de datos de entrada que se suministren.
De hecho, esto ya se está poniendo en marcha, existiendo varias inteligencias artificiales que son capaces de ponerle nombre y apellidos a un diagnóstico, comparar los resultados de una prueba con otros y plantear la situación de cada persona para poder aplicar medicina personalizada. En relación con esto, Hirasawa y colaboradores entrenaron una red neuronal con 13.548 endoscopias de cáncer gástrico y para evaluar su precisión de diagnóstico, se analizaron otras 2296 endoscopias en 47 segundos, diagnosticando correctamente 71 de 77 lesiones por cáncer gástrico, es decir, un 98.6% de acierto.
Si bien es cierto que las inteligencias artificiales son imparables en el campo del diagnóstico al llegar donde no llega un facultativo, aún hay mucho por recorrer ya que una IA pierde el factor humano de poder pasar consulta a un paciente y la psique robótica está aún muy lejos de ser una realidad.
Lo que si es cierto, es que la computación biológica forma parte de la solución a muchos problemas que golpean nuestra sociedad actual, ya que, se podrían fabricar microprocesadores orgánicos y fácilmente biodegradables si quien hace todas las operaciones lógicas es una población de E.coli y no un trozo de silicio y otros materiales contaminantes. ¿Será posible en un futuro mandarle un mensaje de texto a tus amigos a través de una bacteria? ¿Soñó Turing con poder programar en una E.coli?
BIBLIOGRAFÍA
1. Ray, T.S., Evolution, ecology and optimization of digital organisms. 1992, Citeseer.
2. Adleman, L., Molecular computation of solutions to combinatorial problems. Science, 1994. 266(5187): p. 1021-1024.
3. Lenski, R.E., et al., The evolutionary origin of complex features. Nature, 2003. 423(6936): p. 139-44.
4. Baumgardner, J., et al., Solving a Hamiltonian Path Problem with a bacterial computer. Journal of Biological Engineering, 2009. 3(1): p. 11.
5. Green, A.A., et al., Complex cellular logic computation using ribocomputing devices. Nature, 2017. 548(7665): p. 117-121.
6. Hirasawa, T., et al., Application of artificial intelligence using a convolutional neural network for detecting gastric cancer in endoscopic images. Gastric Cancer, 2018. 21(4): p. 653-660.