![]()
Los orígenes de la Bioinformática (II) Las macromoléculas llevan información
El concepto de que las proteínas (que a fin de cuentas son macromoléculas) contienen información que está codificada en forma de secuencias lineales de aminoácidos lleva mucho tiempo totalmente aceptado por la comunidad científica, y es la piedra angular de toda la bioinformática clásica. Pero esto no fue siempre así, teniendo que remontarnos a los años posteriores a la Segunda Guerra Mundial para encontrar las primeras pruebas empíricas de esta idea. El bioquímico Emil Smith (que no lo debéis confundir con Temple Smith, del algoritmo Smith & Waterman) describió este periodo como «heroico» para la bioquímica de proteínas. El periodo heroico comprendería desde 1945 a 1955, cuando fue publicada la secuencia completa de la insulina gracias al esfuerzo de Frederick Sanger (sí, el que da nombre al Sanger Centre) y sus colaboradores.
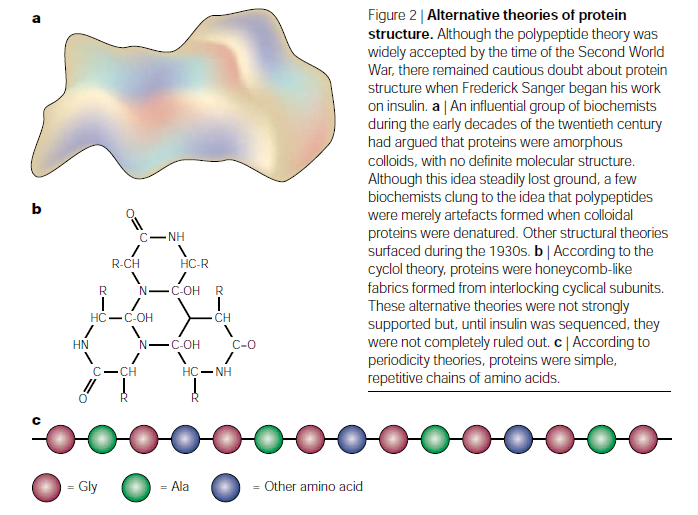
Resumiendo mucho la historia, Frederick Sanger tomó como base el postulado (ahora teoría) sobre polipéptidos en la estructura de las proteínas. Este postulado, formulado inicialmente en 1902, generó mucho escepticismo en la comunidad científica, siendo más aceptadas las hipótesis alternativas (que en su época parecían más creibles). Algunas de ellas están reflejadas en la figura que he puesto. Una de ellas postulaba que las proteínas eran una especie de coloides amorfos, sin estructura definida, y que los polipéptidos se generaban al desnaturalizarse esos coloides. Otra promovía la idea de que las proteínas tenían forma de mallas con estructuras de celdas de colmena, tan típicas del anillo de benceno y los compuestos aromáticos. Y la última de la figura refleja una idea similar, pero no igual, en la que se consideraba la linealidad de la proteína incluso a nivel estructural, donde se consideraba que era una macromolécula periódica compuesta por repeticiones de de cadenas de aminoácidos.
Las técnicas experimentales habían mejorado ostensiblemente en las décadas de 1930 y 1940, pero antes del trabajo del equipo de Frederick Sanger no se sabía apenas nada sobre la posición específica o el orden de cada aminoácido en la proteína, o de su estructura. La resolución y publicación de la secuencia de aminoácidos de la insulina permitió descartar todas las hipótesis erróneas y comprobar a la idea básica de que las proteínas están compuestas de aminoácidos, que esos aminoácidos tienen un orden lineal (secuencia o estructura 1D), y que esos aminoácidos en ese orden específico determinan la estructura de la proteína (estructuras 2D y 3D). El trabajo experimental que condujo a la obtención de la secuencia de la insulina (de sólo 51 aminoácidos) fue bastante arduo, porque requería de químicos muy experimentados en la degradación de las proteínas fueran capaces de determinar de forma muy precisa en qué estado de degradación se encontraban las muestras, e ir controlando esa degradación.
Pero al mismo tiempo otros bioquímicos estaban desarrollando métodos más refinados, como la reacción de degradación de Edman, que permitió ir quitando de una cadena peptídica pequeña uno a uno sus aminoácidos, de forma secuencial. Esta nueva técnica permitió semiautomatizar todo el proceso, lo cuál fue una revolución para el mundo de la biología molecular. Por ejemplo, con esta nueva técnica la secuenciación de la ribonucleasa (realizada por el equipo coordinado por Stanford Moore y William Stein del Instituo Rockefeller), una proteína de 124 aminoácidos, llevó la mitad de tiempo que la obtención de la secuencia de la insulina.
A finales de la década de 1960 Pehr Edman consiguió automatizar por completo todo el proceso con su «sequenator», lo cuál hizo que muchos grupos de biología molecular hicieran crecer la cada vez mayor biblioteca de secuencias de proteínas. Sin estos primeros esfuerzos y entonces nuevas técnicas experimentales habría sido muy difícil que nacieran las bases de datos de secuencias, o que se hubiera si quiera soñado en proyectos de secuenciación del genoma completo de un organismo. Y sin ello la bioinformática, la biología molecular y muchas otras disciplinas relacionadas con las ciencias de la vida posiblemente serían muy diferentes…