![]()
TopHat 2: arreglos para algunos fallos técnicos
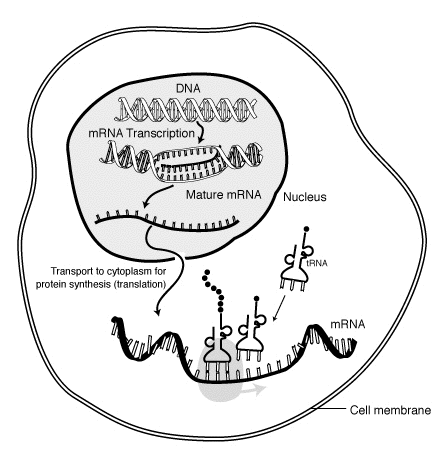
Una parte de la bioinformática actual gira en torno a los análisis de datos de experimentos de ultrasecuenciación: ChIP-Seq, FAIRE-Seq y DNase-Seq, exome sequecing, RNA-Seq, … Una de las herramientas es TopHat (en el momento de escribir esta entrada iba por la versión 2.0.10), que se usa en los experimentos de RNA-Seq para identificar splice junctions tipo exón-exón… Para entender qué es esto de los splice junctions tipo exón-exón, primero hay que recordar cómo funciona la síntesis de proteínas con estos esquemas de la Wikipedia inglesa:
Cuando una proteína del código genético de la célula va a ser producida por la maquinaria celular, lo primero que se hace es, dentro del núcleo, copiar el código genético correspondiente a los exones que representan a esa proteína, descartando normalmente otros exones alternativos del mismo gen (y de los demás genes). Todos esos exones son puestos de forma consecutiva en un ARN mensajero, que es el que sale del núcleo de la célula con esa información y viaja por el citoplasma hasta llegar a un ribosoma. Pero los procesos biológicos encargados de la composición del ARN mensajero pueden fallar, por ejemplo juntando exones correspondientes a proteínas de distintos genes. Estos procesos pueden estar alterados por defectos genéticos, y por diversas enfermedades, como por ejemplo todos los tipos de cáncer.
Los experimentos de RNA-Seq tratan de capturar la información sobre parte del transcriptoma (que sería el conjunto de todos los posibles ARN mensajeros), justo aquéllos que se encuentran en uso en las muestras usadas para el experimento de RNA-Seq. TopHat usa un genoma de referencia relacionado con los datos a analizar (por ejemplo, el genoma humano), información sobre los exones conocidos y a qué genes corresponden (proveniente de UCSC Genome Browser o Ensembl), y los datos a analizar como tal (del orden de decenas de GB).
TopHat, como casi todas las herramientas y pipelines de análisis del mundo NGS, está en continuo desarrollo y es bastante complejo (está escrito en Python 2.7 y C++), con lo que es inevitable que contenga fallos técnicos. Los datos de un experimento de RNA-Seq que tuvo que analizar una compañera de trabajo han tenido la «suerte» de ser capaz de disparar tres de estos fallos técnicos.
- Segmentation fault en segment_juncs: Como se puede ver en esta entrada de un foro de Google Groups, es un fallo existente de antes. La suerte es que en esa entrada vienen las pistas de cómo arreglar dicho fallo en el código fuente, en el fichero src/segment_juncs.cpp .
- AttributeError: 'NoneType' object has no attribute 'split' de tophat al relanzar un trabajo terminado abruptamente: tophat viene con la posibilidad de reiniciar trabajos de análisis que hayan terminado de forma abrupta, desde el punto en el que haya fallado. Pero ese mecanismo tiene fallos… Para los que programéis en otros lenguajes de programación, pero no en Python, ‘NoneType’ es la clase de los ‘None’, el equivalente a ‘undef’ de Perl, ‘nil’ de Ruby, ‘NULL’ de C y C++, ‘undefined’ de JavaScript, etc… También hubo suerte, porque en esta otra entrada del mismo foro de Google Groups, un usuario propone un arreglo que, aunque no sea muy ortodoxo ni correcto, hace que tophat continúe.
- Segmentation fault en tophat_reports: Aunque este fallo está más o menos documentado en el mismo foro de Google Groups, nadie propone un arreglo. Pero investigando un poco aquí, descubrí que el fallo se encuentra en el fichero src/tophat_reports.cpp, por una razón similar a la descrita en el fallo de segment_juncs.
Actualización
Para que os sea más fácil arreglar estos problemas si os topáis con ellos, he puesto los parches derivados de los arreglos en un repositorio de GitHub: https://github.com/inab/tophat2-patches
Actualización 2
Hemos encontrado para nuestro caso la causa principal de los dos Segmentation fault. Había discrepancias entre el genoma de referencia y sus índices de bowtie, que se solucionaron ejecutando de vuelta bowtie-build.