![]()
Los mapas del cólera
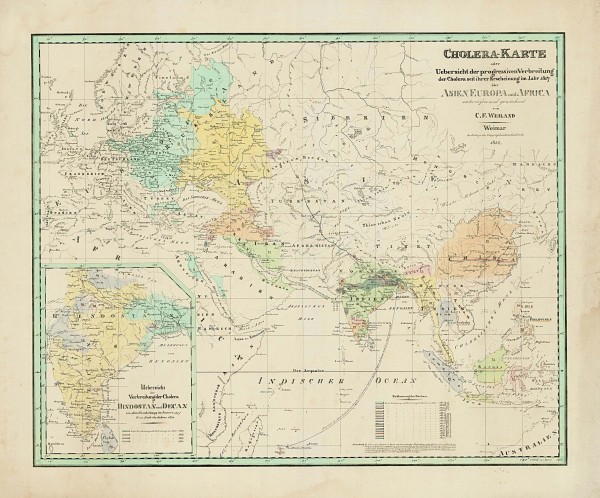
La utilización de mapas como herramientas en epidemiología empezó en el siglo XIX con la llegada a Europa de la segunda pandemia de cólera. Aunque las causas de la enfermedad aún no se conocían, y se creía que la enfermedad la producían «miasmas» que se transmitían por el aire, los mapas publicados en 1831 y 1832 (como el que se muestra en la figura, o los que se muestran en (Koch, 2014)) indicaban que la epidemia no surgía aleatoriamente en uno u otro lugar, sino que progresaba siguiendo rutas precisas que coincidían con campañas militares o rutas comerciales, es decir que se transmitía de un lugar a otro siguiendo los movimientos de grupos de viajeros. En 1854 John Snow, que no creía en los «miasmas», representaba los casos de cólera que se estaban produciendo en ese momento sobre un plano de Londres y conseguía identificar un foco local de la epidemia (comentado por Miguel aquí). Desde entonces la elaboración de mapas epidemiológicos se ha ido enriqueciendo con la recogida sistemática de datos, la informatización, y la utilización de Sistemas de Información Geográfica. La incorporación de información sobre serotipos ó genotipos añadió una nueva capa de complejidad, y permitió trazar las rutas seguidas por diferentes clones de un mismo patógeno. Ya en este siglo, la secuenciación de genomas completos abre el campo de la filogeografía genómica, que aporta un aumento importante a la resolución de los mapas en lo que se refiere a la información genética.
El cólera es una infección intestinal producida por algunos subtipos (los serogrupos O1 y O139) de la especie Vibrio cholerae, una enterobacteria que vive en medios acuáticos y se transmite por la ingesta de agua o alimentos contaminados. En la mayoría de los casos se trata de una infección leve y autolimitada, aunque hasta en un 20% de los casos puede ser grave, y puede producir la muerte por deshidratación si no se trata (para más información aquí). Pero es una infección que puede tratarse, y que puede prevenirse. Y en este sentido, el cólera es un reflejo de las desigualdades sociales en el mundo, las poblaciones más afectadas son las más pobres y vulnerables.
En octubre de 2017 la OMS lanzó un programa internacional cuyo objetivo es reducir en un 90% las muertes por cólera para el año 2030. Las herramientas para llevarlo a cabo existen, y no son especialmente caras ni sofisticadas. Se necesitan acciones coordinadas a diferentes niveles para un conseguir un diagnóstico rápido, un manejo adecuado de los pacientes, una gestión correcta de los residuos generados, el saneamiento de los focos contaminados, y la organización de campañas de vacunación y de información. Y es importante subrayar que existen vacunas orales baratas y eficaces que la OMS pone a disposición de los países que las necesiten.
El cólera se conoce desde hace siglos, sin embargo, la primera pandemia bien documentada es la que dio lugar, a principios del siglo XIX, a los mapas comentados. Desde entonces se han registrado ocho grandes pandemias, de las que las dos últimas aún están activas. La séptima pandemia se inició en 1961, y desde entonces se ha mantenido en forma de múltiples ondas epidémicas. Uno de los países afectados en los últimos años es Yemen, que está sufriendo desde 2016 uno de los peores brotes de cólera conocidos, con más de un millón de casos y más de 2.000 muertes registradas. Para caracterizar en detalle el clon o clones responsables de este brote, un grupo internacional liderado por el Institut Pasteur y el Wellcome Sanger Institute ha secuenciado los genomas completos de 42 cepas de V. cholerae obtenidas de pacientes infectados en tres áreas (gobernaciones) diferentes del país, más los de otras 74 cepas obtenidas en países del sudeste asiático, Medio Oriente y centro y este de África (Weill, 2019). Todas ellas pertenecían al clon O1 El Tor (serogrupo O1, serotipo Ogawa, biotipo El Tor), responsable de la séptima pandemia. Las secuencias se añadieron a una colección de más de 1000 secuencias de genomas de V. cholerae analizadas anteriormente por el mismo grupo (Weill, 2017), y se realizó un análisis filogenético detallado. Aunque la epidemia en Yemen se ha producido en dos oleadas (2016 y 2017), todas las muestras pertenecían al mismo grupo, un sólo clon que habría llegado a Yemen a principios de 2016. A su vez este entronca con clon detectado por primera vez en 2006 en el sudeste asiático, y está más relacionado con los brotes ocurridos durante la última década en los países del centro y este de África (Kenia, Tanzania y Uganda) que con los del Medio Oriente (Irán e Iraq). Es decir, que se trata de un clon reciente que llegó a Yemen desde el sudeste asiático pasando por África.
Como ya sugerían los primeros mapas, el cólera se mueve con los viajeros, sean emigrantes, soldados, comerciantes, voluntarios o turistas, pero genera epidemias donde las condiciones locales son favorables. El clon que está produciendo el cólera en Yemen no parece ser especialmente virulento, además ha perdido varios genes de resistencia a antibióticos, y es más sensible que el clon original. Y sin embargo la situación en Yemen está fuera de control. Como ya se vio en Haití después del terremoto de 2010 son las condiciones locales las que determinan la magnitud de la epidemia. Y la situación en Yemen es la de un país en guerra, con infraestructuras destrozadas, una población debilitada y altamente susceptible, y una capacidad de respuesta institucional muy limitada.
La secuenciación y la filogeografía genómica aportan resolución genética suficiente para trazar con detalle las rutas de diseminación de los patógenos. Teniendo en cuenta los precios y las capacidades de las tecnologías actuales, es viable hoy hacer el seguimiento de cualquier brote epidémico con alta resolución y en tiempo real. El objetivo es identificar las rutas de transmisión y cortarlas, adelantándose y previniendo antes de que se produzcan brotes que pueden llegar a ser incontrolables.
REFERENCIAS
Koch T. 1831: the map that launched the idea of global health. Int J Epidemiol. 2014;43(4):1014-20. PubMed PMID: 25237691
Weill FX, et al. Genomic insights into the 2016-2017 cholera epidemic in Yemen. Nature. 2019 Jan;565(7738):230-233. doi: 10.1038/s41586-018-0818-3. PubMed PMID: 30602788.
Weill FX, et al. Genomic history of the seventh pandemic of cholera in Africa. Science. 2017;358(6364):785-789. doi: 10.1126/science.aad5901. PubMed PMID: 29123067.

Parece interesante como es que John Snow, el pionero de la epidemiologÍa traza una ruta siguiendo la epidemia del cólera con forme aparecen casos nuevos y las características en común que tienen estos pacientes en su alimentación e higiene. En aquel tiempo no se conocía exactamente como es que de una ciudad, la enfermedad aparecía en otra; este texto deja muy claro que es por ingerir alimentos o agua contaminados.
Años después se han hecho grandes avances microbiológicos para convatir esta enfermedad, como la creación de la vacuna.