![]()
Dividirse adecuadamente para multiplicarse bien
Con seguridad las publicaciones de Craig Venter no pasan desapercibidas, si no por su resultado si por su objetivo, reformular el genoma de un microbio, ya de por sí reducido, para convertirlo en lo mínimo capaz de mantenerse vivo. Lleva ya un par de décadas en el empeño y podemos decir que sin haberlo logrado por completo sí que se va acercando. Cada vez los genomas que consigue excluyen genes que no parecen imprescindibles para que la célula sobreviva en el laboratorio en donde es mantenida con un mimo exquisito.
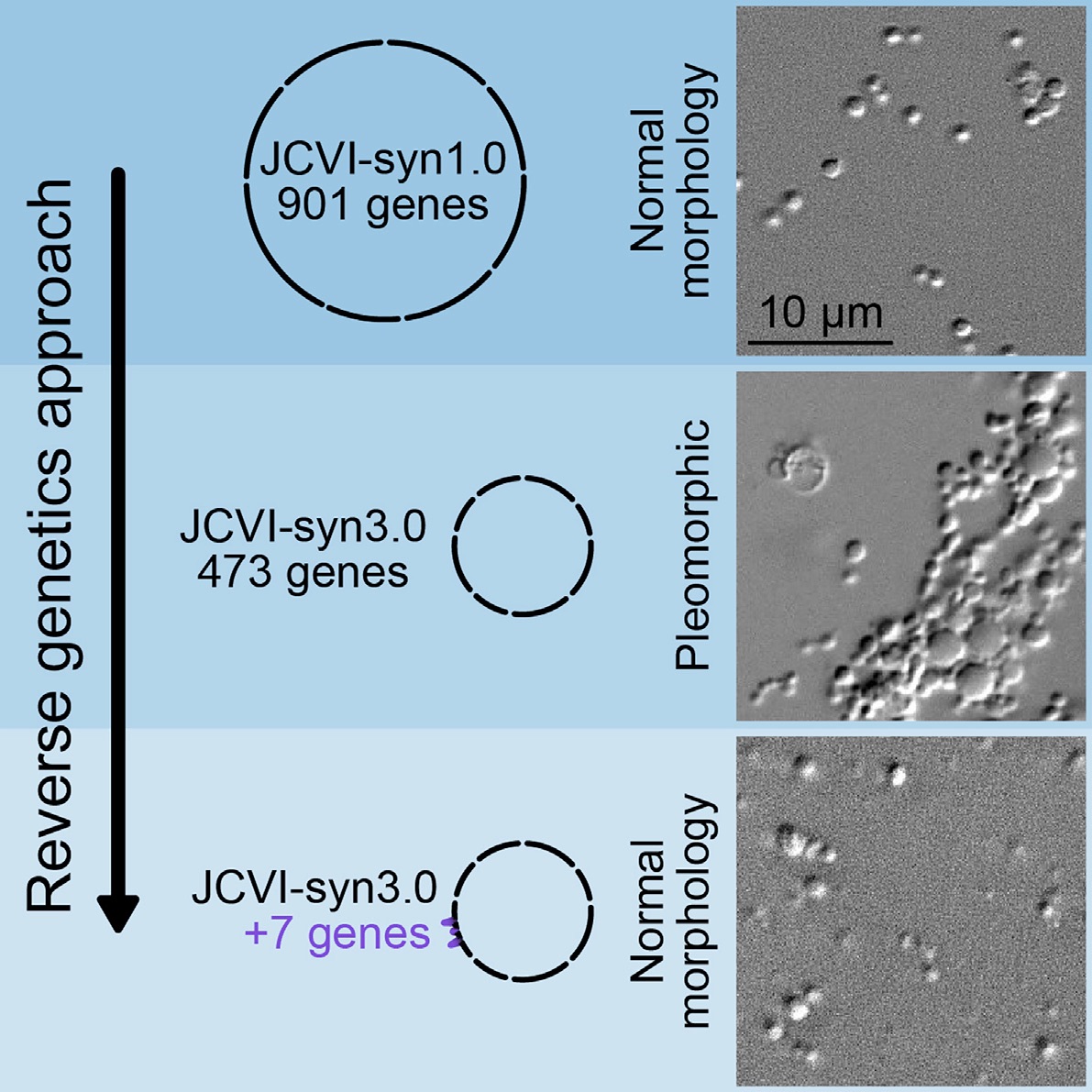
Comprobación de que hay al menos siete genes que son necesarios y suficientes para devolver la capacidad de dividirse a un Mycoplasma que como resultado de una deleción solo se podía multiplicar por fragmentación. La población de la estirpe delecionada, imagen en la fila del centro, al no dividirse regularmente, tiene una gran variación en el tamaño y en la forma de sus individuos. La integración de esos siete genes, a la estirpe que aparece en la fila de abajo, restituye el tamaño y la forma de la estirpe inicial normal, en la fila de arriba. La imagen muestra las poblaciones de cada estirpe observada mediante una celda de cultivo que permite el crecimiento continuo y su visualización al microscopio, un desarrollo técnico que ha facilitado mucho la realización del trabajo.
Quizás lo más atractivo de las publicaciones de Venter es que nos obligan a pensar sobre lo que es una célula viva. Excluimos del argumento a los virus, que no son células y que no pueden vivir fuera de una célula a la que parasitan. La última publicación de Venter ha intentado llegar a un genoma mínimo que parece mantener en parte la función esencial de la división, al menos permite al organismo rediseñado mantener un tamaño próximo al de la bacteria original.
Las Mycoplasma son las bacterias de menor tamaño y carecen de peptidoglicano, el polimero responsable de la rigidez de la pared bacteriana por lo que no adoptan una forma fija, son pleomorfas. Por la misma razón son además resistentes a antibióticos como la penicilina. En cierta medida recuerdan a las llamadas formas L que adoptan algunas bacterias cuando se les elimina la pared celular. En trabajos anteriores del grupo habían ido reduciendo el genoma de un Mycoplasma, ya de por si pequeño si se compara con bacterias como Escherichia coli, la bacteria más estudiada como modelo para definir los mecanismos de división. Esos estudios no iban más allá de comprobar si las estirpes resultantes eran viables, pero uno de los puntos que no habían considerado antes era el cómo podía mantenerse la división celular. Las bacterias muy grandes pueden crecer y sobrevivir pero cuanto mayor tamaño tienen peor les va. Lo mismo que las formas L, al no tener una pared rígida la fragmentación de las células muy alargadas puede dar lugar a individuos de menor tamaño, lo que podría en cierta medida revitalizar a la población.
En el trabajo actual, publicado recientemente en la revista Cell, Venter y su equipo han realizado un abordaje de genética inversa para identificar algunos genes que intervienen en la división celular de Mycoplasma. Partieron hace años de una estirpe que por su morfología redondeada y su tamaño pequeño y bastante uniforme determinaron que se divide de forma conmensurada con su crecimiento.
Eliminando varios genes para minimizar el genoma el proceso se descontrola y se observa que la estirpe resultante es pleomórfica, hay muchas células muy voluminosas, redondas y alargadas, lo que sugiere que no hay coordinación entre el crecimiento y la división. Integrando a continuación en el genoma un fragmento con 19 de los genes eliminados en el paso anterior se recupera el control, las bacterias resultantes vuelven a ser de tamaño reducido, son en su mayoría redondeadas y la población es bastante uniforme. De entre los 19 genes encuentran que 7 de ellos son suficientes para restaurar la división y los consiguen identificar.
En los bacilos como E. coli y Bacillus subtilis muchos genes que intervienen en la formación del septo de división, como ftsZ, se localizan juntos en un agrupamiento llamado dcw. También en ese agrupamiento se localizan varios genes que codifican enzimas para la síntesis del peptidoglicano.
La proteína FtsZ es una de las tres que en E. coli forman el proto-anillo que interviene en la formación del septo de división. Parece lógico que ftsZ sea uno de los siete genes identificados. FtsZ no se asocia por sí misma a la membrana, por lo que también parece lógico que el gen que codifica FtsA, una de las otras dos proteínas del proto-anillo que junto a ZipA sirve para anclar FtsZ a la membrana sea otro de los siete genes identificados. También entre ellos se identificó el gen sepF que codifica una de las proteínas necesarias para la división y que en otras bacterias como Mycobacterium puede servir para el anclaje de FtsZ. Sigue dentro de la lógica el que como Mycoplasma no tiene peptidoglicano ninguno de los otros genes se relacione con enzimas que lo sinteticen.
De los otros cuatro genes hay dos, mraW y mraZ que si se asocian al agrupamiento dcw en E. coli pero no se sabe que intervengan en la división. Tampoco a rpmF, que codifica una proteína de los ribosomas, ni al gen que nos queda que no tiene nombre y codifica una posible hidrolasa de función desconocida se les puede encontrar una conexión sencilla con la división.
Cómo ocurre la división celular en la estirpe que contiene todos los genes que devuelven a Mycoplasma la viabilidad y la morfología silvestre es algo que no queda esclarecido en la publicación. Se echa en falta una comparación más minuciosa de los resultados del trabajo con los obtenidos mediante el análisis molecular tradicional del ciclo de división en bacterias modelo, tanto E. coli como B. subtilis y Streptococcus. Quizás pudiera ser por eso que a los autores les hayan podido pasar inadvertidas algunas correlaciones.
Las formas L, como dijimos carentes también de peptidoglicano no se dividen formando un septo, sino que lo pueden hacer, al aumentar su masa, por fragmentación, algo que podía ser una propiedad intrínseca de las membranas biológicas cuando forman vesículas y aumenta su volumen. También cuando existe un exceso de la proteína ZipA la membrana de E. coli responde formando pliegues e invaginaciones incluso cuando está recubierta de la capa rígida de peptidoglicano. Podría postularse que el peptidoglicano fue una adquisición de una protocélula que se multiplicaba por fragmentación y en la que su interior sólo contenía macromoléculas a la misma concentración que su medio ambiente. Añadirle una cubierta rígida permitiría a las bacterias ser más eficaces al poder concentrar su citoplasma y así maximizar las interacciones precisas para regular mejor sus procesos fisiológicos. A la par, la presencia del peptidoglicano tuvo que ir acompañada por la adquisición de mecanismos complejos de septación incluyendo el ensamblaje de FtsZ en un proto-anillo. Desde el punto de vista filogenético es de todas formas más plausible que Mycoplasma sea una degeneración de una bacteria y no un ancestro. La presencia en su genoma de algunos de los elementos del agrupamiento dcw así lo atestiguaría.
REFERENCIA:
Pelletier et al., 2021, Cell 184, 1–11. April 29, 2021 Published by Elsevier Inc. enlace