![]()
«Motifs»: los ladrillos de una red compleja
Hace unos 10 años, el laboratorio de Uri Alon introdujo el concepto de «network motifs» (Nature Genetics 31, 2002): patrones de interacción recurrentes en una red compleja que aparecen en un número significativamente más alto que el esperado en una red aleatoria equivalente (aquella con igual número de nodos y enlaces distribuidos éstos de forma aleatoria). Por ejemplo, imaginemos una red dirigida, aquella con interacciones asimétricas como las que tienen lugar entre las distintas especies de un ecosistema, donde X -> Y representa que X (depredador) se alimenta de Y (presa).
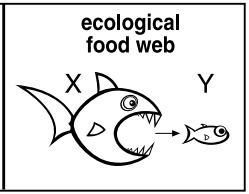
Si consideramos todas las posibles formas de conectar 3 nodos en una red dirigida, nos encontramos con las siguientes 13 combinaciones:
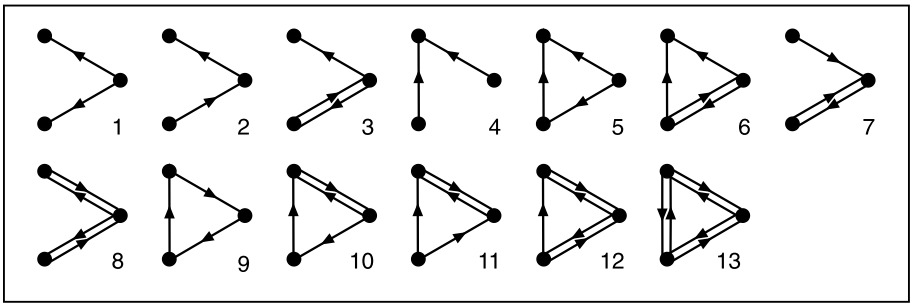
Los «motifs» representarían los ladrillos que se van seleccionando durante el crecimiento de la red para realizar una determinada función. Así, en redes tróficas, la combinación número 2 (X->Y->Z, permite que haya un flujo de energía de abajo a arriba en la cadena alimentaria) es mucho más frecuente que cualquier otro patrón de interacción de 3 nodos, mientras que en las redes de regulación genética o redes neuronales es el número 5 («feed-forward loop«) el motivo más abundante (un circuito que sólo procesaría señales de entrada procendentes del entorno que sean persistentes en el tiempo).
Detectar y entender estos circuitos elementales que componen una red compleja puede ayudar a comprender el funcionamiento global de la red y establecer clases de equivalencia de redes.
