Las características que afectan a la flexibilidad del ARN de doble cadena no se habían estudiado en detalle hasta ahora
El plegamiento de las moléculas de ADN tiene una gran importancia de cara a la accesibilidad a la información genética y la expresión de los genes en el momento adecuado. Numerosos estudios han analizado cómo la secuencia del ADN afecta a la estructura y flexibilidad de la molécula. El ejemplo más representativo lo encontramos en unas secuencias llamadas A-tracts, que regulan el plegamiento del genoma en el interior de la célula, facilitando que el ADN pueda enrollarse alrededor de las histonas para conformar los nucleosomas.
En nuestro organismo, además del ADN, el ARN también puede formar dobles cadenas. Estas moléculas participan entre otras cosas en la regulación génica o en la respuesta a infecciones virales. Sin embargo, al contrario del ADN, se desconocía si la secuencia del ARN de doble hebra afecta a su estructura y flexibilidad. Ahora, científicos del Consejo Superior de Investigaciones Científicas en el Centro Nacional de Biotecnología (CNB- CSIC) y de la Universidad Autónoma de Madrid (UAM) han identificado secuencias concretas dentro del ARN (que han denominado AU-tracts) que permiten aumentar la curvatura estas moléculas. Estos resultados se publican en la revista Nucleic Acids Research.
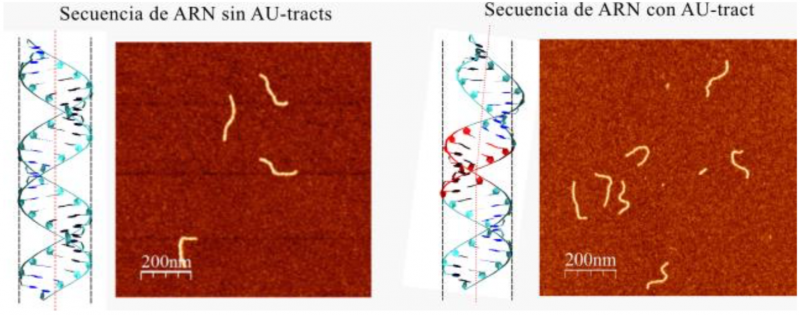
Simulación de ARN sin AU-tracts encajado en un cilindro vertical e imagen en AFM de moléculas sin AU- tracts (izquierda) y con AU-tracts curvado (se sale del cilindro) e imagen en AFM de moléculas con AU- tracts (derecha). Alberto Marín-González y Mikel Marín-Baquero (CNB-CSIC).
Alberto Marín, investigador del CNB-CSIC y primer autor del trabajo explica: “realizando comparaciones computacionales de una variedad de moléculas de ARN con distintas secuencias hemos observado que aquellas que contenían regiones concretas con bases de adenina y uracilo seguidos (AU-tracts) se curvaban, a diferencia de las moléculas que no incorporaban estas secuencias. Fue un resultado totalmente inesperado”. Para confirmar estos resultados, los investigadores han diseñado moléculas de ARN con secuencias AU-tracts y analizado su disposición utilizando un microscopio de fuerzas atómicas (AFM). “De esta forma, hemos comprobado que las secuencias AU-tracts en las moléculas individuales de ARN de doble hebra modulan la curvatura del ARN”, detalla Fernando Moreno-Herrero, uno de los investigadores que han liderado el trabajo.
Este descubrimiento abre muchas preguntas a explorar en el futuro. Según Mikel Marín, coautor del trabajo “queda por entender qué papel juegan estas estructuras de ARN en el interior de la célula y cómo regulan las interacciones entre ARN y proteínas”. Más allá de su papel en biología, las estructuras de los AU-tracts abren la puerta a diseñar moléculas de ARN con determinadas propiedades estructurales. “Nuestro descubrimiento podría contribuir a impulsar la nanotecnología basada en ARN. Este es un campo relativamente reciente, pero muy prometedor. Tiene por objetivo construir estructuras tridimensionales de ARN en la nanoescala con aplicaciones en nanomedicina, como la administración de fármacos o el diagnóstico mediante biosensores; o en nanotecnología, por ejemplo permitiendo la fabricación de estructuras que podrían servir como andamio en ingeniería de tejidos”.
Más información:
Double Stranded RNA bending by AU-tract sequences. A Marín-González, C Aicart- Ramos, M Marín-Baquero, A Martín-González, M Suomalainen, A Kannan, JG Vilhena, UF Greber, F Moreno-Herrero and R Pérez. Nucleic Acids Research, 2020 doi: 10.1093/nar/gkaa1128

