![]()
¿Es posible reconstruir la división celular sin célula?
autor: Miguel Vicente

Oscilación de MinCD provocada por MinE. La predicción del modelo publicada hace siete años. En rojo MinE, en verde MinCD.
Meinhardt and de Boer . 2001. Proc. Natl. Acad. Sci. USA 98: 14202-14207.
Persecución molecular
El trabajo del grupo alemán ha consistido en demostrar que las proteínas MinD y MinE pueden reproducir fuera de la célula las oscilaciones que realizan en su interior desde un extremo al otro. MinD es un potenciador de MinC, un inhibidor de la división celular que impide el funcionamiento de FtsZ. Los investigadores han mezclado las dos proteínas sobre una doble capa de lípidos (como si fuese una membrana celular) colocada encima de una lámina de mica. Para distinguirlas han marcado una fracción de cada una de ellas con compuestos que producen fluorescencia al iluminarlos con luz ultravioleta, han elegido el verde para MinD y el rojo para MinE. De lo que se sabemos sobre cómo funcionan MinD y MinE en la célula podemos decir que MinE debería ir tras MinD empujándolo desde el lugar que ocupa en la membrana hacia el extremo. Esto es exactamente lo que las dos proteínas hacen en el ensayo sin células.
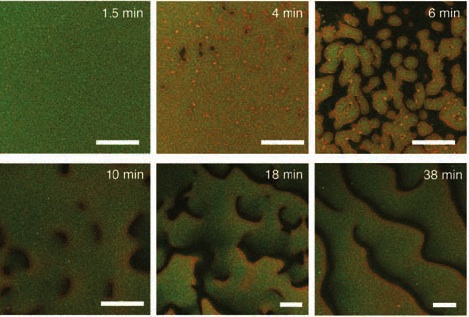
MinD se despega del soporte y forma ondas cuando se añade MinE. Los números marcan el tiempo transcurrido desde la adición de MinE a una superficie recubierta uniformemente por MinD. La barra marca una longitud de 50 µm.
Cuando la preparación solo contiene MinD se observa una lámina continua de proteína recubriendo a la membrana, al añadir MinE la uniformidad se rompe y se crean ondas de MinD separadas una de otra por MinE. El proceso necesita ATP, una de las formas de suministrar energía a las reacciones biológicas.
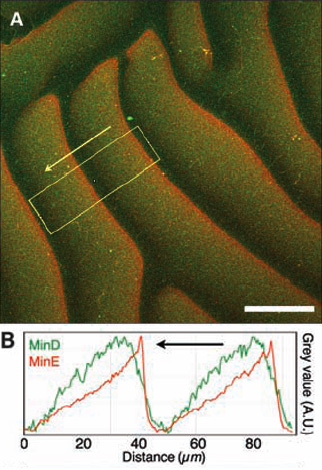
MinE persigue a MinD. En el recuadro A se ve en rojo la fluorescencia de MinE, que empuja a la fluorescencia verde de MinD. En el recuadro B se ha cuantificado la fluorescencia verde y roja del recuadro señalado en A. La flecha señala la dirección del desplazamiento de la onda de fluorescencia verde. La escala marca una longitud de 50 µm.
Apretarse el cinturón
FtsZ, la proteína con la que ha trabajado el equipo estadounidense, es precisamente la que sufre los efectos del complejo MinCD (formado por MinC y MinD), y es la que inicia la formación del anillo de división en el centro de la célula, el lugar en donde, debido a la oscilación de uno a otro extremo, menos tiempo permanece MinCD. Se sabe que FtsZ forma polímeros siempre que no haya MinCD. FtsZ no puede unirse por sí misma a la membrana de la bacteria, se cree que lo hace por medio de otras dos, FtsA y ZipA. Lo que han hecho los investigadores para simplificar el sistema es pegarle a FtsZ un segmento de la proteína MinD, justo el responsable de que ésta se una por sí sola a la membrana. También le han pegado una proteína fluorescente (en este caso amarilla) para poder visualizarla. Esta quimera, muy diferente de la proteína original, no es capaz de dividir a la bacteria, pero sí se puede colocar en el interior de liposomas (unas vesículas que reproducen un recinto parecido al interior de la célula) y se incorpora a su membrana. Suministrándole GTP, la molécula que en este caso aporta la energía, se observa que la FtsZ quimérica forma polímeros en forma de anillo, como haría la FtsZ original en el centro de la célula. Lo más interesante es que estos anillos artificiales, que solo se forman sobre superficies con una determinada curvatura, son capaces de tirar de la membrana hacia el interior, se produce así un adelgazamiento del liposoma en la zona donde está el anillo que se interpreta como la constricción que marca el sitio de división en la bacteria.
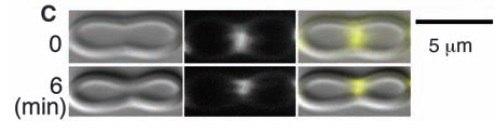
FtsZ consigue estrechar el lugar del liposoma donde se localiza. La imagen de abajo se tomó seis minutos después que la de arriba. Las imágenes de la izquierda, obtenidas por contraste de fase muestran el contormo del liposoma. Las del centro son la fluorescencia de la quimera FtsZ, y las de la derecha la imagen compuesta en la que la fluorescencia se ha coloreado de amarillo
Biología Sintética
Los dos trabajos nos demuestran que se puede reconstruir, sin necesidad de que haya células, parte de la maquinaria de división de las bacterias. Además de ser interesante para aplicarlo en ensayos de nuevos fármacos antibacterianos permitirá comprobar si lo que sabemos sobre el proceso de división celular es correcto en todos sus aspectos o si por el contrario intervienen elementos adicionales que no conocemos. Nos dicen además que en el control de los procesos celulares complejos juegan un importante papel las propiedades intrínsecas de algunos de sus componentes. Así la propiedad de MinE de empujar a MinD de un lado a otro puede determinar sin más cuál es el centro de la célula por donde ha de localizarse el tabique que separe a las dos hijas. El mecanismo de polimerización de FtsZ puede, por su parte, estrangular la membrana de la célula y contribuir al proceso de constricción. Todo esto va a servir para dentro de poco sintetizar la maquinaria en su totalidad y conseguir que se dividan, como si fuesen células, estructuras artificiales que no lo son.
REFERENCIAS
M. Loose, E. Fischer-Friedrich, J. Ries, K. Kruse, and P. Schwille 2008. Spatial regulators for bacterial cell division self-organize into surface waves in vitro. Science 320: 789-792.
M. Osawa, D.E. Anderson, and H.P. Erickson. 2008. Reconstitution of contractile FtsZ rings in liposomes. Science 320: 792-794.

Foro del día en madridiario

¡Qué bonito!
Saludos
http://curiosidadesdelamicrobiologia.blogspot.com/
Buen dia saludos, tengo una inquietud quiza alguien me pueda ayudar es posible que al poder moldear celulas puedo alterar la masa de un objeto? a tal manera que yo pueda contralar? gracias mi email: alejandro_k1989@hotmail.com