![]()
Deconstruyendo las bacterias: nueva cocina en la Microbiología
autor: Miguel Vicente
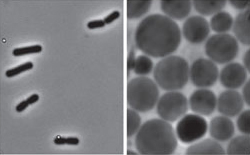
Comparación de células normales de Bacillus subtilis a la izaquierda con las respectivas formas L a la derecha.
Un grupo de investigadores de la Universidad de Newcastle ha logrado producir formas L de B. subtilis y al observarlas se ha encontrado con alguna sorpresa, estos engendros no solo pueden crecer, incluso se multiplican y lo hacen sin recurrir a los mecanismos que las bacterias normales utilizan. Se construyeron formas L de B. subtilis conteniendo una fusión de FtsZ con la proteína fluorescente de medusa (la GFP) y se observó que aunque se llegaban a formaban arcos fluorescentes, nunca llegaban a completar un anillo, la estructura de FtsZ que interviene en la división bacteriana. El paso siguiente fue construir una estirpe en la que se puede forzar la pérdida del gen ftsZ en determinadas condiciones. Lo sorprendente fue encontrar que esas formas L no solo crecían, sino que podían proliferar.
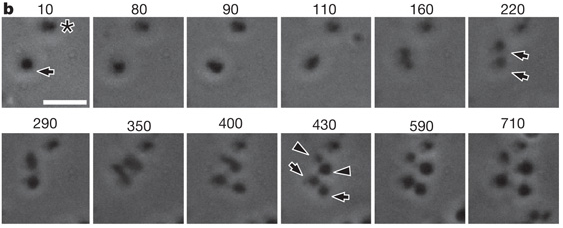
Secuencia de imágenes en las que se observa la partición de formas L de Bacillus subtilis.
Hace poco más de un año autores de Francia y España publicaron que algo parecido a las formas L se podía obtener a partir de Escherichia coli, pero en este caso su multiplicación dependía de una cantidad residual, un 7%, de peptidpoglicano, ya que la inhibición de la proteína PBP3, que sintetiza el peptidoglicano del septo bloquea por completo su división. ¿A qué se puede deber esta discrepancia? En primer lugar a que la receta para producir las formas L usada por los dos grupos es diferente, en el caso de Bacillus se obtienen bloqueando por completo la síntesis del peptidoglicano sustituyendo la expresión natural del operón murE, que contiene genes para codificar varias enzimas necesarias para sintetizar sus precursores, y haciendo que se puedan dejar de transcribir a voluntad, y a la vez bloqueando la síntesis del precursor bactoprenol. Por el contrario lo que se hizo en E. coli fue bloquear la producción de peptidoglicano añadiendo cefsulodina, un antibiótico que bloquea dos de las enzimas, las llamadas PBPs 1A y 1B, que intervienen en el ensamblaje de los precursores del petidoglicano. Pero también debe tenerse en cuenta que nos encontramos ante dos tipos de bacteria muy diferentes con respecto a la composición de su pared, Bacillus es una Gram-positiva, con una pared más sencilla compuesta por una membrana única rodeada de una gruesa capa de peptidoglicano. E. coli por su parte es una Gram-negativa, con dos membranas entre las que hay una capa delgada de peptidoglicano.
Nos quedan algunos interrogantes, uno es cómo se la arreglan las formas L de Bacillus para efectuar la constricción de su membrana. Los autores británicos piensan que quizás intervengan otras moléculas del citoesqueleto bacteriano, como MreB, homóloga de la Actina, y que el proceso pueda funcionar de forma parecida a la emisión de seudópodos. ¿Era este un mecanismo de división en los ancestros de las bacterias?
REFERENCIAS
M. Leaver, P. Domínguez-Cuevas, J. M. Coxhead, R. A. Daniel and J. Errington. 2009. Life without a wall or division machine in Bacillus subtilis. Nature 457: 849-853.
D. Joseleau-Petit, JC. Liébart, JA. Ayala, and R. D’Ari. 2007. Unstable Escherichia coli L-forms revisited: growth requires peptidoglycan synthesis. J Bacteriol 189: 6512–6520.

Hola
¡Jo, que interesante! Ahora resulta que es MreB, la prima de FtsA, la que puede ser "la sal" para es plato que conocemos como "división celular".
Un abrazo
Muchas gracias por la información sobre las Formas L… muy interesante
Saludos
[…] otros investigadores como Jeff Errington, han logrado que otras bacterias, del género Bacillus, cuando se les elimina el peptidoglicano se propaguen por fragmentación. Necesitan también ser mimadas en unas condiciones que solo […]