![]()
Conoce a tu enemigo
Conoce a tu enemigo y conócete a ti mismo; en cien batallas, nunca saldrás derrotado (Sun Tzu, El Arte de la Guerra).
Entre enero de 2020 y abril de 2021 se han depositado en la base de datos GISAID más de un millón de secuencias de SARS-CoV-2, el coronavirus responsable de la pandemia de COVID-19. A éstas hay que añadir otras ≈260 000 en Genbank. Aunque la mitad de las secuencias de GISAID son de Reino Unido y Estados Unidos, en total son 172 países los que han aportado secuencias. Esto supone un esfuerzo global sin precedentes, sobre todo teniendo en cuenta que no existe ningún proyecto ni directriz global, sino que se trata de cientos de iniciativas independientes de gobiernos y grupos de investigación de todo el mundo en un movimiento de base (bottom-up) en toda regla. En España hay grupos que han secuenciado por su cuenta y con recursos propios, pero además el Instituto de Salud Carlos III ha financiado un proyecto de alcance nacional, SeqCOVID, que ha producido excelentes resultados, y más recientemente el Ministerio de Sanidad y las Comunidades Autónomas están desarrollando un plan para integrar la secuenciación genómica en la vigilancia epidemiológica del SARS-CoV-2.
La secuenciación del genoma completo del virus es necesaria para entender la pandemia y su evolución. Para clasificar o distinguir especies o variedades de animales y plantas se utilizan características como el tamaño, el color y la forma. Con los virus los caracteres morfológicos no son suficientes para diferenciar variantes de una especie, a veces ni siquiera diferentes especies de un mismo género. Sin embargo, igual que los animales y las plantas, los genomas de los virus contienen información genética, que es necesaria para el virus y su ciclo vital, pero que además es útil para los investigadores porque permite diferenciar unos linajes de otros y trazar la evolución del virus.
Desde que comenzó la pandemia la población de SARS-CoV-2 se ha expandido por todo el mundo acumulando mutaciones a un ritmo de 2-3 por genoma y por mes (que es un ritmo típico de los coronavirus). Al estudiar un período de tiempo tan breve las diferencias que se pueden observar entre los distintos linajes son pocas, por eso no basta con secuenciar algún pequeño fragmento del genoma viral, es necesario secuenciar el genoma completo para recoger toda la información posible. El genoma del SARS-CoV-2 tiene casi 30 000 bases. Con ese tamaño es impracticable y económicamente inviable secuenciar cientos o miles de muestras con el método clásico de secuenciación de Sanger. Afortunadamente, durante la última década los métodos de secuenciación masiva (o NGS, next-generation sequencing) se han hecho accesibles a laboratorios medianos y pequeños, esto ha favorecido la participación de centenares de laboratorios de todo el mundo en la secuenciación del virus a gran escala. A su vez, la pandemia está dando un impulso muy grande a la implantación de estos métodos más allá de los grandes laboratorios de referencia.
Cabe recordar que la secuenciación de ácidos nucleicos, sean genomas o sean fragmentos menores, es una tecnología muy potente porque produce información digital objetiva, independiente del laboratorio o la persona que la realice y fácil de compartir mediante bases de datos. El método de secuenciación de Sanger, que todavía tiene su utilidad y sus aplicaciones, es un monumento a la bioquímica y la biofísica del siglo XX. Sin embargo, los métodos modernos de NGS, que son varios y muy diferentes entre sí, incorporan toda la potencia de la electrónica e informática actuales que los confieren capacidades y posibilidades realmente asombrosas e inimaginables hace unos pocos años.
Desde que empezó la pandemia la población del virus ha generado múltiples linajes que se han dispersado por todo el mundo, cada uno acumulando mutaciones diferentes y divergiendo de la secuencia original. Las secuencias genómicas trazan esa historia (Figura 1).
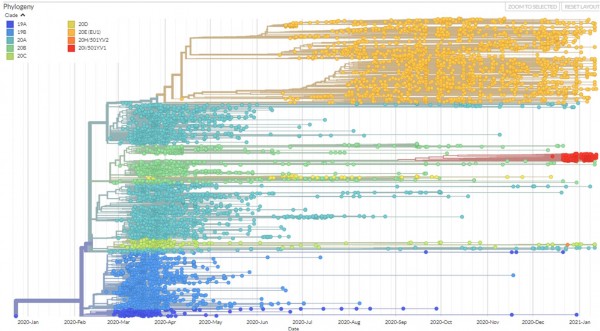
Las secuencias muestran también convergencia evolutiva, la aparición de las mismas mutaciones de manera independiente en diferentes linajes señalando la ocurrencia de procesos de selección natural (en respuesta a la presión inmunológica, por ejemplo). Esto va a ser muy importante para seguir la evolución del virus en la era post-vacunal y detectar variantes capaces de escapar a la respuesta inmunológica producida por las vacunas. Por otra parte, con una orientación más clínica, se está estudiando el impacto de las diferentes variantes, sus frecuencias de transmisión y sus patrones de infección. Además, la secuenciación se está utilizando a escala local para caracterizar y delimitar brotes epidémicos y a escala de pacientes individuales para confirmar los casos de reinfección. Y cuando por fin se desarrollen agentes antivirales habrá que monitorizar sus actividades sobre las diferentes variantes y estudiar el posible escape de variantes con mutaciones de que confieran resistencia.
En definitiva, para conocer al virus secuenciamos su genoma.
