![]()

El (cito)Esqueleto de Loki
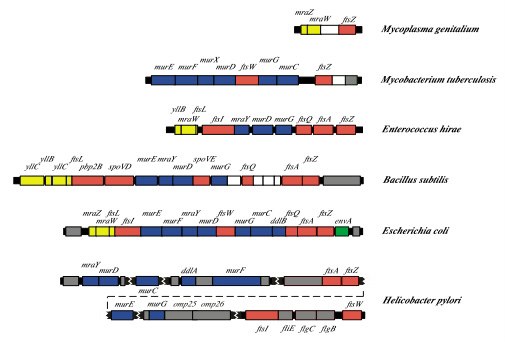
Las arqueas asgardianas no son un nuevo grupo de superheroínas de la Marvel. Son un tipo de microorganismos procariotas descubiertos en el año 2015 en unos sedimentos provenientes de la chimenea hidrotermal conocida como «Castillo de Loki» en las profundidades del Océano Ártico. En el mundo de la microbiología y la evolución, las arqueas pertenecientes al superphylum Asgard son famosas porque se piensa que, hace unos 2.000 millones de años, uno de sus antecesores fue el que consiguió establecer…