![]()
¿Las bacterias son arcaicas o son modernas?: la estructura de los tARNs revela una genealogía sorprendente.
autor: Miguel Vicente

La genealogía de los tARNs. En su conjunto en el panel de arriba ysegún sus dos tipos de estructura en el del centro y abajo. En los trescasos las bacterias aparecen como un linaje joven. Fuente: F-J. Sun andG. Caetano-Anollés, PloS Comp Biol.
Hace relativamente poco tiempo que se formuló la tesis de que los ancestros comunes de todas las células divergieron produciendo tres dominios, arqueas, bacterias y eucariotas, estas últimas con su material genético separado del resto de la célula dentro de un núcleo rodeado de una membrana adicional a la que separa la célula del exterior. Se postuló que arqueas y eucariotas podían compartir ancestros comunes, y que ambas los compartían con las bacterias. Asimismo existen indicios de que las células eucariotas derivan de la simbiosis de una bacteria que en un tiempo se instaló a vivir en el interior de algo parecido a una arquea.
Es difícil aventurar si realmente eso ocurrió así, no en balde estamos hablando de sucesos que bien pudieron ocurrir hace tres mil quinientos millones de años, año más o año menos y que no han dejado huellas fiables en el registro de fósiles que es lo que normalmente se maneja para averiguar la genealogía de bichos más consistentes como los dinosaurios. Por eso esta genealogía se obtuvo estudiando la similitud de algunas secuencias de los ARNs del ribosoma, en concreto del llamado 16s ARN, en los seres vivos actuales. Los ribosomas son complejas fábricas moleculares que en el interior de las células se dedican a ensamblar las proteínas a partir de sus componentes, los aminoácidos. Los ribosomas más antiguos podrían ser más parecidos a los de las bacterias actuales que a los de nuestras células.
Evitar el estallido
Hay algunos indicios indicando que las primeras células debían parecerse a una bacteria Gram-positiva, las bacterias que solo tienen una membrana. Entre ellos la organización en el mismo lugar del cromosoma de los genes que codifican las enzimas que catalizan la síntesis de la pared bacteriana junto a los que forman el septo que divide a las hijas en el momento de la reproducción. Es precisamente en la composición de la pared y de la membrana en donde las diferencias entre bacterias y arqueas son muy apreciables. Y esto no es trivial, porque de la presencia de una pared rígida depende que las bacterias no estallen por la presión de su contenido.
Sin embargo hay ahora otras observaciones que apuntan a un origen más moderno de las bacterias, y curiosamente al origen más arcaico de los virus, que son parásitos incapaces de reproducirse por sí solos y a los que generalmente se considera descendientes de las células que parasitan. Estos nuevos datos se han obtenido analizando la estructura de 571 tARNs de diversos genomas.
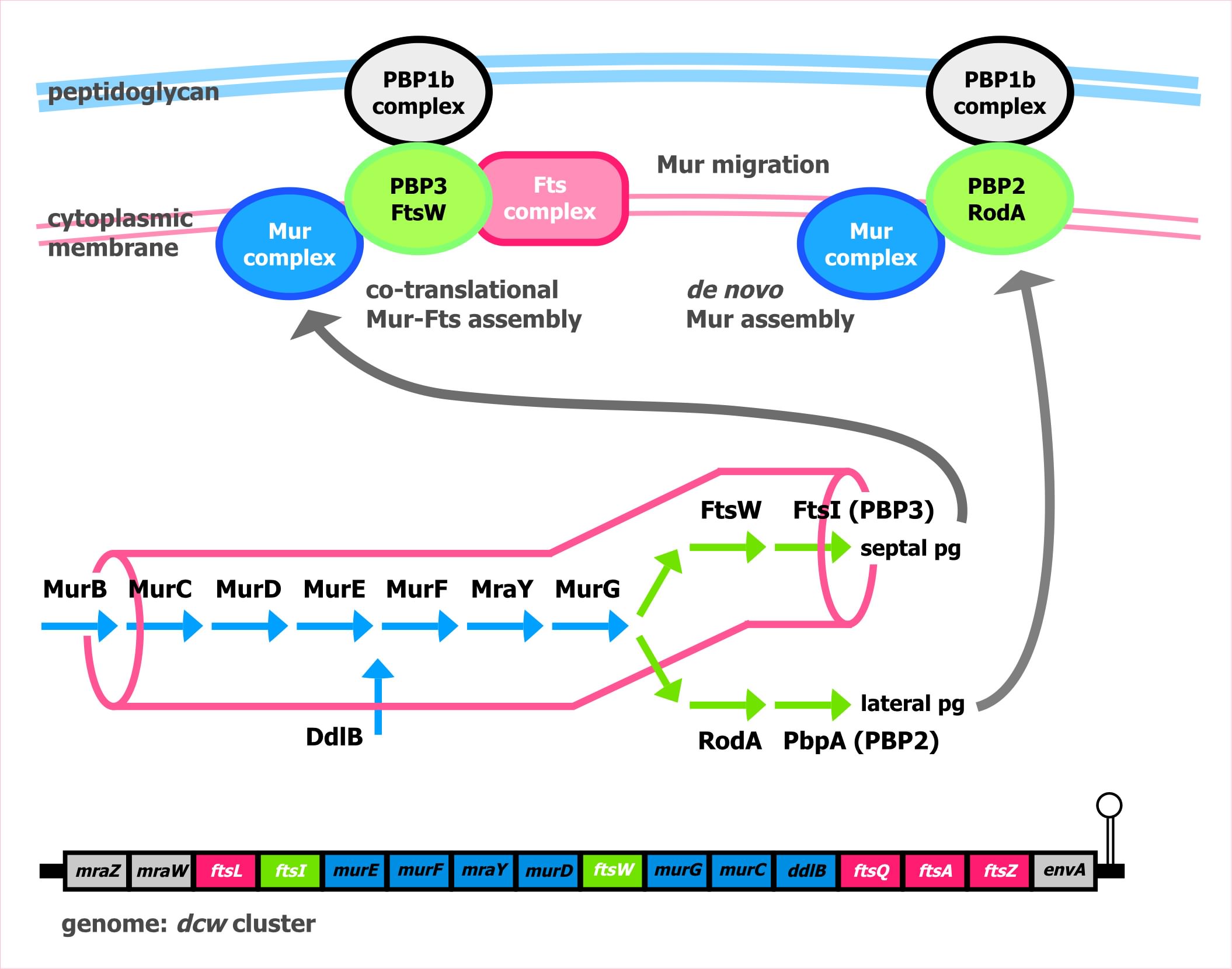
El grupo génico dcw de Escherichia coli. La agrupación de los genes que codifican enzimas que intervienen en la síntesis de la pared junto a los que codifican proteínas de anillo de división puede conferir ventajas en las bacterias de forma bacilar, lo que se ha denominado «canalización genómica«. Fuente: Vicente et al. 2006. Septum enlightenment: the assembly of the bacterial division proteins. J. Bacteriol. 188: 19-27.
Fábrica de proteínas
Para sintetizar una proteína, que en definitiva va a ser la molécula con actividad biológica dentro de la célula, se necesita convertir la información de los genes desde el ADN hasta una cadena de aminoácidos que luego se pliega para formar la molécula activa. En este proceso interviene un ARN llamado mensajero que traslada la información desde el ADN al ribosoma. En el ribosoma, donde se ha colocado el mensajero, los tARNs interpretan la información del lenguaje que usan los genes, 64 tripletes (palabras) diferentes, y la traducen al lenguaje de unos veinte distintos aminoácidos que se combinan siguiendo esa pauta impresa en el ADN para formar las proteínas. Para obtener su genealogía, que en un principio usando los métodos clásicos de análisis no parecía arrojar ningún resultado, se ha recurrido a forzar un poco la aplicación del proceso de identificación del parentesco. La constricción ha consistido en no dejar que cada dato de un tARN caiga en el lugar más adecuado a su estructura, sino forzar a los datos a pertenecer a uno u otro de los dominios conocidos, sea virus, bacteria, arquea o eucariota. De esta manera las estructuras que parecen más antiguas son las de los tARNs de arqueas, y sorprendentemente los virus. Y la última sorpresa es que las bacterias se colocan en una etapa más cercana al presente que las células eucariotas.
Evolución modular
Si descartamos que la genealogía obtenida sea errónea, cosa que probarán o refutarán investigaciones futuras, especialmente cuando se determinen las estructuras de los tARNAs de más especies, muy posiblemente lo que los resultados nos indican es que los organismos actuales, incluyendo en ellos a los virus, han surgido por evolución de diversos módulos. Así por un lado los genes de pared y división han podido adoptar una arquitectura que facilita la proliferación de algunas bacterias, mientras por otro los genes de los tARNs han adoptado varias estructuras, cada una de ellas se incorporó a cada dominio en un determinado momento a partir de una reserva de estructuras comunes. Al menos en el caso de los virus esta opción parece casi obligatoria pues mal puede establecerse un parásito sin que exista previamente el organismo al que parasita.
REFERENCIA
F-J. Sun and G. Caetano-Anollés. 2008. Evolutionary patterns in the sequence and structure of transfer RNA: early origins of archaea and viruses. PLoS Comput Biol 4(3): e1000018. doi:10.1371/journal.pcbi.1000018

según su aporte cual es la antiguedad de las bacterias en millones de años