![]()
Si quieres mejorar una levadura, acelera la evolución
Como suele decirse, primero se hace en procariotas y después en eucariotas. En el año 2014 se consiguió hacer un cromosoma artificial de levadura. Fue un proyecto que involucró desde estudiantes de grado a laboratorios punteros en Biología Molecular. La Facultad de Medicina de la Universidad John Hopkins organizó un cursillo llamado «Build-A-Genome» en el que los estudiantes fueron creando y uniendo secuencias cortas de 70 nucleótidos para formar bloques de 750 pares de bases que luego fueron unidos por investigadores de otros laboratorios para ser ensamblados en grandes pedazos que una vez introducidos en una levadura, fueron ensamblados en un único cromosoma.
Tres años después el avance ha sido 5 veces más grande pues se han conseguido introducir 5 cromosomas artificiales. En un reciente número de la revista Science se publican siete artículos describiendo los diferentes aspectos de este avance tecnológico. En la imagen de la portada de la revista podemos contemplar una reconstrucción en 3D de la organización de los cromosomas artificiales (color dorado) y naturales (color blanco). Eso significa que estamos un poco más cerca de llegar al mismo punto que se consiguió con los micoplasmas cuando se sustituyó todo su genoma por un genoma artificial. Pero aquí es un poco más difícil porque hay que sustituir los 16 cromosomas de un la levadura por cromosomas de diseño. Y todo eso ¿para qué sirve?
La idea final es eliminar todos los transposones, elementos repetitivos, recodificar codones stop, introducir información que permita una manipulación genética más fácil… sin que la levadura sufra ningún problema en su eficacia biológica. Ya vimos en la entrada anterior escrita por Miguel Vicente, que toquetear los genes que codifican los ribosomas es como meterse a hacer reformas del hogar: algunas cosas como la cocina es mejor no cambiarlas de sitio. Hay que tener en cuenta que el genoma de los seres vivos está sometido a una gran diversidad de contigencias evolutivas en forma de mutaciones y recombinaciones que crean variabilidad en la población. Y la Selección Natural ejerce su acción sobre esa variabilidad. Hace varios milenios el ser humano comenzó a domesticar a la levadura Saccharomyces cerevisiae y gracias a ella pudo elaborar diversos productos como la hidromiel, el pan, la cerveza o el vino. La domesticación significó que ser humano ejerció una nueva presión de selección sobre el genoma de la levadura. Pero esa nueva presión de selección se estaba ejerciendo sobre algo que la Naturaleza había creado. Y una cosa que tenemos muy claro en Biología es que a veces la Evolución comete errores. Pues bien, construir una levadura con cromosomas artificiales sería el gran paso en la domesticación de este microorganismo pues nos permitiría manipular sus capacidades, conservar aquellas que nos parecen más interesantes e incluso eliminar esos errores evolutivos tan molestos.
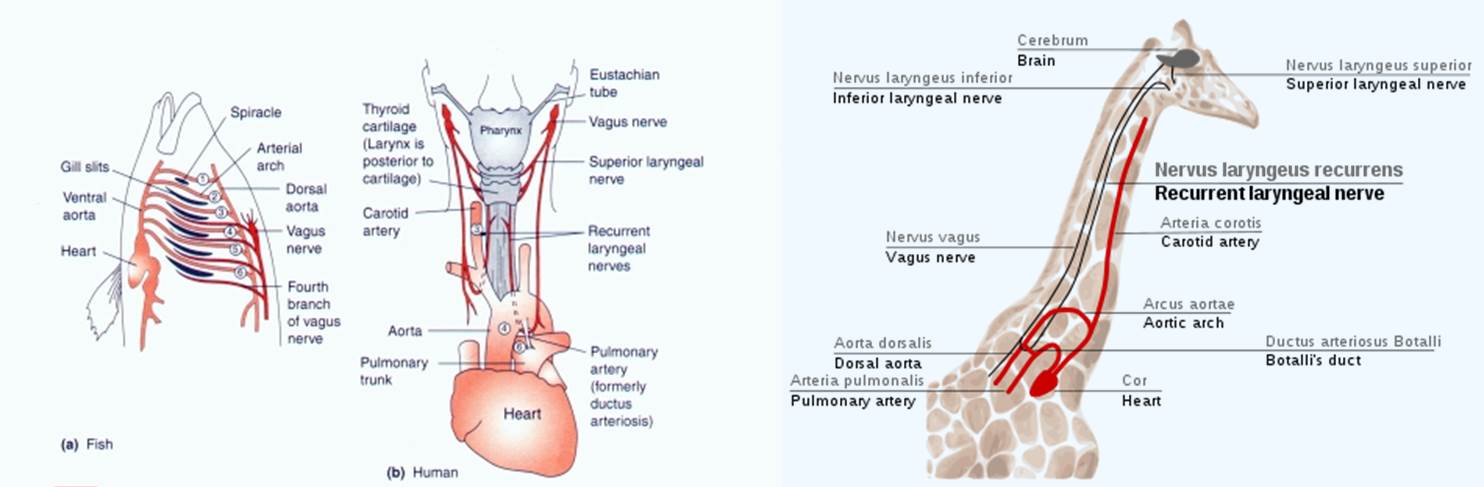
Vamos a explicar primero lo que es un «error evolutivo». El ejemplo clásico de los libros de texto es el del nervio laríngeo recurrente. Este nervio es la cuarta ramificación del nervio vago, un nervio craneal que va desde el cerebro a la laringe. Pero lo curioso es que este nervio no sigue un camino «recto». Lo que hace es salir del cerebro, baja por el cuello casi hasta el corazón, rodea la aorta y vuelve hacia arriba por el cuello hasta la laringe. En los humanos eso significa aumentar por cuatro la distancia que debe de recorrer el nervio (además de embrollar el «cableado» de nervios y vasos sanguíneos). Pero si nos fijamos en las jirafas el problema de diseño es realmente peliagudo porque el nervio, en lugar de recorrer unos 20 centímetros desde el cerebro a la laringe, tiene que recorrer una distancia de ¡4 metros! El «error evolutivo» se explica cuando uno considera que los mamíferos evolucionamos a partir de los peces hace muchísimo tiempo. Nuestro ancestro no tenía cuello y los nervios lo que inervaban era un arco branquial. Pero con el tiempo (y la acción de la Evolución) parte de las branquias se transformaron en la laringe y en la aorta, y como el nervio no se podía cambiar de posición, lo único que se pudo hacer fue alargar su recorrido.
Pues con las células pasa lo mismo. Uno de los «errores evolutivos» tiene que ver con la gran cantidad de DNA repetitivo que existe en el genoma de la levadura (y en el de todos los eucariotas). El origen de ese DNA es variado: integración de provirus o de transposones, duplicaciones genéticas, etc. El problema es que ese DNA también es replicado y por lo tanto la célula gasta energía y recursos en mantenerlo. Bueno, pues ya que se manipulan y se sintetizan artificialmente unos cromosomas nuevos, se aprovecha la ocasión y se eliminan todas esas secuencias de DNA repetitivo.
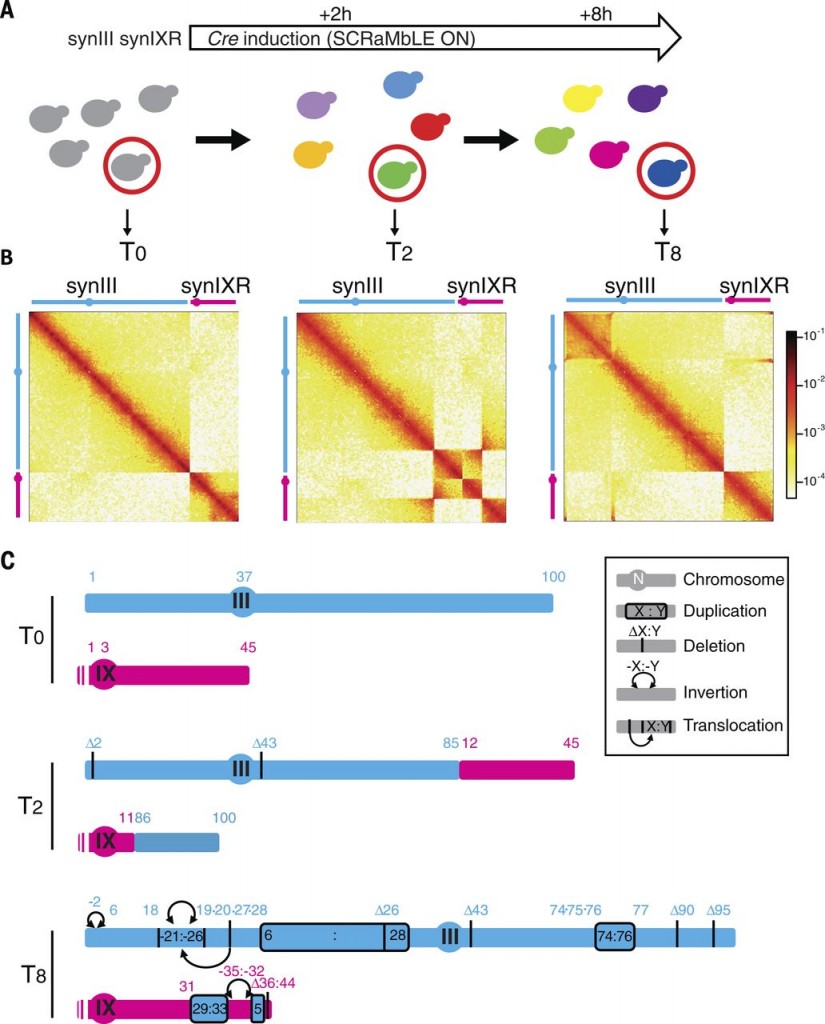
Pero los científicos no se han conformado con esto. Han introducido en los cromosomas sintéticos cientos de secuencias especiales denominadas loxPsym que permiten modificar o reorganizar dichos cromosomas a voluntad. Lo han denominado SCRaMbLE por las siglas en inglés de (Synthetic Chromosome Rearrangement and Modification by LoxP-mediated Evolution) que traducido significa «Modificación y Reorganización Cromosómica debida a Evolución Mediada por LoxP» aunque en cristiano lo de SCRaMbLE significaría algo así como «revoltijo». Y es que precisamente eso es lo que hace el sistema Cre-Lox: cortar y pegar genes hasta que hace un revuelto genético. Como se muestra en la figura inferior, esto permite que se pueda inducir una «evolución acelerada» de la levadura ya sea mediante duplicación, deleción o reorganización de la información contenida en los cromosomas sintéticos.
Parece que todo indica a que podremos mejorar el diseño de la levadura para que haga muchas más cosas de interés, siempre y cuando en la reforma no le «toquemos las narices«.