![]()
Los fósiles en las bacterias están vivos… y algunos controlan su proliferación.
Autores: Miguel Vicente y Jesús Mingorance
Averiguar cómo ha sido la evolución de los seres vivos actuales, qué relaciones tienen entre ellos y de qué ancestros descienden no es tarea sencilla. Es preciso conocerlos al detalle y también disponer de los restos de sus antepasados, por ejemplo de toda una “Sima de Atapuerca” de huesos fósiles para reconstruir la de los homínidos más recientes.
¿Cómo podremos reconstruir el linaje, lo que los científicos llaman filogenia, de las bacterias si no tienen huesos? Desde hace tiempo los científicos que han estudiado la filogenia bacteriana han recurrido, en ausencia casi total de fósiles, a estudiar el ADN de las bacterias vivas. Se podría decir que como el ADN se hereda de generación en generación, el genoma de una bacteria viva, donde están sus genes, es como una “sima de Atapuerca” que ha pasado de sus ancestros hasta las bacterias actuales.
Lógicamente para establecer la filogenia solo sirve el análisis de la secuencia de los genes comunes en todas ellas, como ocurre con los genes ribosomales que son imprescindibles para ensamblar los ribosomas, unos complejos moleculares encargados de sintetizar las proteínas en las células de todos los seres vivos. Los virus, que no son células, son un caso aparte.
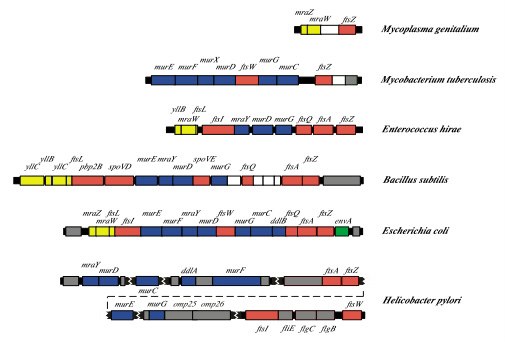
Hace ya años se observó que no solo se ha conservado la secuencia de los genes compartidos, también en algunos casos se ha mantenido el orden en que aparecen dentro del genoma. Esta conservación del orden es más frecuente en los genes que, como los de los ribosomas, tienen un papel estructural y menos en los que lo tienen funcional. Una grata sorpresa ha sido encontrar que se conserva también el orden del agrupamiento dcw, un conjunto de genes de tipo funcional.
El agrupamiento dcw incluye muchos de los genes encargados de que se sintetice la pared bacteriana y del divisoma, que es el conjunto de proteínas imprescindible para que se ejecute con precisión la división de la bacteria. El dcw suministra así información sobre la filogenia de las bacterias como si fuese un registro fósil de los ancestros de las bacterias actuales.
No ha sido fácil concluir que en las bacterias actuales, además de conservarse, los genes del agrupamiento dcw también se han mantenido en el mismo orden. Durante décadas se han aportado datos por varios investigadores, en particular por quienes estudiamos los mecanismos moleculares que forman el divisoma. En el inicio se encontró que en Escherichia coli, cerca de genes como ftsA y ftsZ, que codifican proteínas del divisoma, se localizaba un gen mra que codifica una proteína responsable de la síntesis del peptidoglicano, el componente cuya estructura mantiene la rigidez de la pared bacteriana y evita que las bacterias estallen por la presión a la que está su interior. En trabajos posteriores se encontraron más de esos genes tanto de división como de pared y la genética molecular indicaba que varios se encontraban en la misma zona del cromosoma. Fueron las técnicas de secuenciación del ADN las que finalmente mostraron que ese lugar del genoma de E. coli solo contiene genes relacionados con la división celular y la síntesis de la pared y por eso lo designamos como agrupamiento dcw, acrónimo de división y pared celular (en inglés division and cell wall). La observación bien hubiera podido quedarse así, al menos para mi papel (M. V.) en esta historia, si no hubiese intervenido la afortunada circunstancia de una invitación al ITQB en Oeiras, Portugal, donde trabajaba Adriano Henriques. Él había leído el borrador de un artículo de revisión en el que junto con Juan Ayala proponíamos nombrar al conjunto como dcw (Ayala et al., 1994). Adriano había participado en la redacción de una revisión similar en Bacillus subtilis (Buchanan et al., 1994) y me dijo que en esa bacteria, tan poco parecida a E. coli como un humano a un percebe, los genes equivalentes también estaban agrupados, todos ellos orientados en el mismo sentido y en el mismo orden. A partir de ahí el número de bacterias en las que se iba revelando la existencia del agrupamiento dcw aumentó y fue la colaboración que mantuvimos con Alfonso Valencia y Javier Tamames aplicando técnicas informáticas la que nos permitió comprobar la existencia del agrupamiento en un número mayor de bacterias, todas ellas de forma bacilar (Tamames et al., 2001). Así se llega al presente, en el que un grupo del Instituto Pasteur de París ha analizado el agrupamiento dcw en 1081 genomas pertenecientes a los 102 filos bacterianos (o divisiones) reconocidos actualmente (Megrian et al., 2022). Los genes del agrupamiento se identifican en los genomas de todos los filos, y el agrupamiento se ha encontrado completo en la mayoría (57%), fragmentado en la tercera parte (32%), y disgregado en el 10% de ellos. Más importante, en todos los casos en los que se ha identificado el agrupamiento, el orden de los genes se conserva. A veces algunos genes están ausentes o se encuentran fuera del dcw, pero por otra parte hay genes que se encuentran invariablemente dentro del agrupamiento y siempre en la misma posición relativa.
No se conoce ningún mecanismo que justifique el orden en el que se encuentran los genes dentro del agrupamiento dcw, ni su conservación. No es un operón, no todos los genes se expresan como una unidad de transcripción. Nosotros propusimos en su día la hipótesis de canalización genómica que postulaba que durante la síntesis de las proteínas el orden de los genes podría facilitar el ensamblaje de algunos complejos macromoleculares y coordinar el crecimiento de la pared celular y el del septo de división en las células bacilares (Mingorance et al., 2004). Sin embargo, en el trabajo de Mergian et al., con muchos más genomas y un rango taxonómico más amplio, no se encuentra una asociación entre la estructura del dcw y la forma de las células, descartando, o al menos no confirmando, nuestra hipótesis. Tampoco se encuentra relación con la estructura de la pared celular (una o dos membranas) o la capacidad de formar esporas.
Los autores del trabajo realizan una reconstrucción del contenido y el orden de los genes del dcw y concluyen que el ancestro común a todas las bacterias existentes (LBCA, last bacterial common ancestor) tenía un agrupamiento de dieciséis genes en exactamente el mismo orden en que se encuentran en los genomas actuales, y que las fragmentaciones y pérdidas de genes son caracteres derivados que han ocurrido de manera independiente a lo largo de la historia evolutiva de las bacterias. Esto concuerda plenamente con las conclusiones de otro trabajo similar publicado también este año por otro grupo (Léonard et al., 2022) y sugiere que el dcw puede ser una buena herramienta para reconstruir la filogenia de las bacterias y resolver dudas o ambigüedades observadas con otros marcadores. Por ejemplo, Megrian et al. lo aplican al estudio del filo CPR (Candidate Phyla Radiation), un grupo enigmático, de amplia distribución geográfica pero con muy pocas especies cultivadas, y comprueban que en consonancia con la taxonomía previa el filo está formado por dos subgrupos, y que pertenece al superfilo Terrabacteria (los clásicos gram-positivos monodermos, con membrana celular y sin membrana extena) tal como sugerían algunos estudios de microscopía electrónica.
Si el LCBA, el ancestro de las bacterias actuales, ya tenía un agrupamiento dcw, se trata entonces de una estructura genética muy antigua, mucho más antigua que los animales, las plantas o los microorganismos eucariotas unicelulares. El hecho de que el orden de los genes se haya conservado durante dos o tres mil millones de años indica que, de alguna manera, debe estar implicado en un mecanismo (desconocido) de regulación o coordinación entre el crecimiento y la división de las células. Un mecanismo que no parece ser esencial, puesto que el agrupamiento puede fragmentarse o disgregarse, pero que debe ser lo suficientemente importante como para conservarse total o parcialmente en el 90% de los filos analizados. El dcw es, sin duda, un fósil viviente y una herramienta muy valiosa para reconstruir la historia evolutiva de las bacterias.
Referencias
Ayala J. A., Garrido T., de Pedro M. A. and Vicente M. (1994) Molecular biology of bacterial septation. In: New Comprehensive Biochemistry. Bacterial Cell Wall, vol. 27, pp. 73–101, Hakenbeck R. and Ghuysen J. M. (eds), Elsevier, Amsterdam.
Buchanan C. E., Henriques A. O. and Piggot P. J. (1994) Cell wall changes during bacterial endospore formation. In: New Comprehensive Biochemistry. Bacterial Cell Wall, vol. 27, pp. 167–183, Hakenbeck R. and Ghuysen J. M. (eds), Elsevier, Amsterdam.
Léonard RR, Sauvage E, Lupo V, Perrin A, Sirjacobs D, Charlier P, Kerff F, Baurain D. Was the Last Bacterial Common Ancestor a Monoderm after All? Genes (Basel). 2022 Feb 18;13(2):376. doi: 10.3390/genes13020376. PMID: 35205421; PMCID: PMC8871954.
Megrian D, Taib N, Jaffe AL, Banfield JF, Gribaldo S. Ancient origin and constrained evolution of the division and cell wall gene cluster in Bacteria. Nat Microbiol. 2022 Dec;7(12):2114-2127. doi: 10.1038/s41564-022-01257-y. Epub 2022 Nov 21. PMID: 36411352.
Mingorance J, Tamames J, Vicente M. Genomic channeling in bacterial cell division. J Mol Recognit. 2004 Sep-Oct;17(5):481-7. doi: 10.1002/jmr.718. PMID: 15362108.
Tamames J, González-Moreno M, Mingorance J, Valencia A, Vicente M. Bringing gene order into bacterial shape. Trends Genet. 2001 Mar;17(3):124-6. doi: 10.1016/s0168-9525(00)02212-5. PMID: 11226588.


[…] la anterior entrada del blog Mingo y Miguel nos explicaron la conservación filogenética del agrupamiento dcw, donde se […]