![]()
Fármacos del Suelo en Beneficio de la Humanidad (nuevas técnicas genómicas, e investigación participativa)
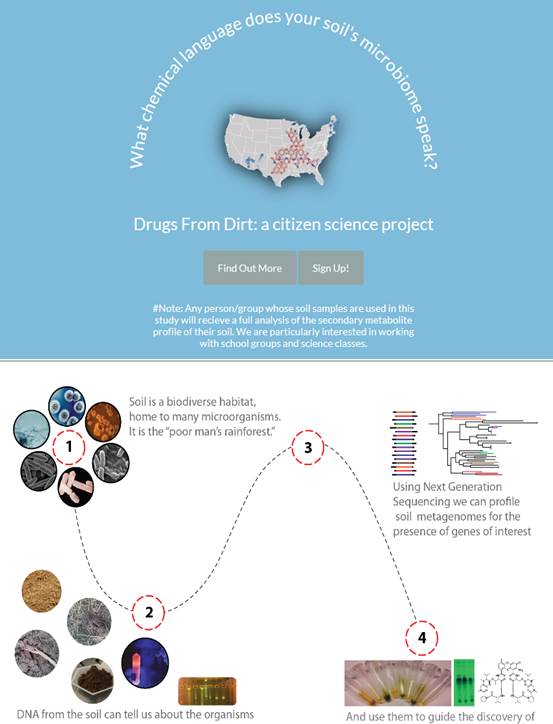
Drugs From Dirt: a citizen science project
Buena parte de los ciudadanos desconocen la gran variedad de fármacos descubiertos en el suelo, así como que estos han salvado cientos o miles de millones de vidas. Gran parte de estas drogas dieron lugar a antibióticos (como por ejemplo la tetraciclina y la vancomicina, etc.). La Medicina tiene una deuda importante a los microorganismos edáficos y especialmente con las bacterias y hongos. Estos pequeños bichitos también han proporcionado o inspirado medicamentos contra el cáncer y terapias inmunosupresoras utilizadas en los trasplantes de órganos. Y todo ello a pesar de que, como os menos narrado en post precedentes, apenas conocemos la diversidad que habita en el medio edáfico. Hasta no hace mucho tiempo, los microbios que habitan en los suelos debían aislarse tras crecer en placas Petri. Sin embargo el problema estriba en que hoy sabemos que la mayor parte de ellos no se desarrollaban en tales medios de cultivo, pasando inadvertidos a los científicos. Sin embargo las actuales técnicas de secuenciamiento genómico permiten ahora detectar genes o grupos de genes que los investigadores pueden identificar como fármacos potenciales, directamente sobre las muestras de tierra. Obviamente, tales sofisticados procedimientos logran detectar enormes cantidades de moléculas, algo que hace una década parecía impensable. La nota de prensa del artículo que analizamos hoy ofrece resultados más que interesantes, y no solo desde el punto de vista farmacológico. Omitiré referirme a los procedimientos utilizados ya que se encuentran más allá de los escasos conocimientos que tengo en tales materias. Abajo podréis consultaros en la nota de prensa y el resumen del trabajo original, publicado en el PNAS. Sin embargo el avance es desorbitante ya que, a día de hoy, la bioprospección de unas pocas muestras de suelo puede dar lugar a identificar una ingente cantidad de compuestos, algunos de los cuales servirán para la producción de nuevos fármacos.
No obstante el trabajo nos informa de muchos más temas. Por ejemplo, Los autores compararon la riqueza y diversidad de genes biosintéticos de 96 microbiomas de los suelos muestreados en una gran variedad de ambientes pertenecientes a todas las regiones del suroeste y noreste de los Estados Unidos. Más aun, como veremos, desean ampliar su colección demandando a los edafólogos interesados y al público en general el muestreo de perfiles de suelos de todos los Estados de la Unión (USA), como manifiestan en su página Web. Se trata pues también de animar al ciudadano a que lleve a cabo la denominada investigación participativa. Más aun los autores del trabajo sesearían llevar a cabo mapas de diversidad genómica alrededor de todo el mundo. Traduciendo literalmente su frase: «The Brady Lab quisiera extender este estudio y espera animar a los científicos y ciudadanos para contribuir a esta iniciativa. El laboratorio ha creado un sitio web: www.drugsfromdirt.org en el que los ciudadanos y edafólogos interesados, después de registrarse, recibirán información acerca de cómo tomar muestras y enviarlas a nuestros laboratorios. El proceso es simple, dice Brady: «Tome una bolsa de sándwich, una cuchara o una espátula, y vierta un par de cucharadas de la bolsa, enviándonoslas después.». Barrunto que si los compuestos que resulten de interés serán después patentados, y hasta los participantes tendrán que pagar para consumirlos cuando los necesiten. Puro capitalismo neoliberal. Pero sigamos.
Los autores compararon la riqueza y diversidad de genes biosintéticos de 96 microbiomas de los suelos muestreados en una gran variedad de ambientes de todas las regiones del suroeste y noroeste de los Estados Unidos.
Ellos encontraron una relación entre el tipo de suelo y el tipo de moléculas que sus microbios residentes tenían la capacidad de producir. Por razones que no entendemos, los suelos áridos resultaron albergar microbios capaces de producir una mayor diversidad de compuestos. Seguidamente tales investigadores comentan: «Esperamos poder ampliar a otras regiones del país y del mundo, para incorporar muchas más muestras con el fin de crear mapas de la diversidad biosintética de los microorganismos del suelo«.
La sentencia en la que se indica una relación entre los tipos de suelos y las sustancias metabólicas segregadas o que atesoraban los microrganismos me resulta intrigante. Hasta el momento, no existen evidencias científicas contundentes para poder afirmar que tipos de suelos distintos atesoran comunidades microbianas idiosincrásicas. Se han publicado artículos que decían corroborar tal correspondencia mientras que en otros se negaba. ¿Cabría pues la posibilidad de que llegaran a obtenerse evidencias más firmes al respecto por esta vía?. Sinceramente permanezco a la espectativa.
Finalmente los autores apuntan a que: “Esperamos poder ampliar nuestro estudio a otras regiones del país y del mundo, para incorporar muchas más muestras con el fin de elaborar mapas de la diversidad biosintética de los microorganismos del suelo”. Ya sabemos que las ciencias del suelo captan escasos fondos en comparación con otras muchas disciplinas científicas. Pero si comparamos las ingentes cantidades de dinero que se invierten en medicina, salud pública e investigación farmacéutica, con la calderilla con la que nos vemos obligados a investigar los edafólogos, las diferencias son abismales. Cuando inicié la andadura en esta bitácora ya os comente en diversos post que la bioprospección de suelos y regolitos debía ser una línea a explorar. Y el estudio así lo sugiere. Si realmente estos investigadores obtienen resultados exitosos el estrecho y sinuoso camino por el que actualmente transcurre la biología, genética y bioquímica del suelo actual se convertiría en una enorme autopista. Esperemos que así sea.
Os dejo ya con la información comentada, aunque escrita en suajili……
Juan José Ibáñez
Searching for drugs in dirt: Researchers call on citizen scientists
Date: May 16, 2014; Source: Rockefeller University
Summary: Microbes are not only a rich source of disease, but also a rich source of medicines, and experts think many life-saving compounds produced by as-yet-unnamed bacteria are awaiting discovery. But they don’t always give up their secrets easily. Researchers must know where to look to find promising bacteria, and how to get them to grow in the lab, the traditional route to identifying potentially valuable molecules they produce.
Getting dirty. Researchers, including postdoc Zach Charlop-Powers (above), are looking for antibiotics and other natural products by sequencing the genetic material found in soil.
Microbes are not only a rich source of disease, but also a rich source of medicines, and experts think many life-saving compounds produced by as-yet-unnamed bacteria are awaiting discovery. But they don’t always give up their secrets easily. Researchers must know where to look to find promising bacteria, and how to get them to grow in the lab, the traditional route to identifying potentially valuable molecules they produce.
Researchers in Sean Brady’s Laboratory of Genetically Encoded Small Molecules are working on a way around these roadblocks. By using genomic sequencing technology, they can investigate the organisms that live in habitats like soil without having to grow the microbes in the lab. They are using this information to map out the location of gene clusters they believe may encode novel antibiotics, and, with help from citizen scientists around the country, they are hoping to process soil samples from areas they would never be able to visit on their own.
In a preliminary effort, Brady’s lab has surveyed nearly 100 soil samples from two U.S. regions, looking for genetic sequences that encode certain molecule-making abilities. «We hope to expand to other regions of the country and the world, to incorporate many more samples in order to create maps of the biosynthetic diversity of soil microbes,» says Zachary Charlop-Powers, a postdoc in the lab. «These maps could help guide drug discovery by identifying variants on known bacterial genes that might be part of a gene cluster encoding a new antibiotic.»
Medicine already owes a major debt to microbes, particularly bacteria. These tiny organisms have produced or inspired many antibiotics, from tetracycline to vancomycin, as well as cancer-fighting drugs and immune system-suppressing therapies used for organ transplants. These bacterial natural products are part of the organisms’ chemical defense system and these molecules have historically been isolated from the broth of bacteria grown in the laboratory.
«However, genetic evidence hints there are many, many more bacteria out there that we may not be able to grow,» Brady says. «And they should be an equally rich source of useful natural products. We have been developing genetic tools to help us look for new chemistry by looking at the genes used to synthesize these natural products.»
For the past five years, Brady’s lab has been sequencing and shifting through DNA obtained directly from soil to identify potentially useful genes, which the researchers then transplant into more-laboratory friendly bugs.
Charlop-Powers, Brady and colleagues recently published the first geographical survey intended to speed this discovery process in the Proceedings of the National Academy of Sciences. For this study, they focused on genes responsible for producing two important families of biologically active molecules: nonribosomal peptides and polyketides. These families include most of the therapeutic molecules isolated from cultured bacteria, but in spite of this diversity, the underlying genetic architecture remains constant. In these families, repetitive genetic domains generate molecules in an assembly line-like fashion that evolution has frequently retooled.
In DNA from 96 soil samples collected for the survey, the researchers looked at two of these domains to get a sense for the diversity and richness of microbes capable of producing compounds these families. They found a link between the type of soil and the sorts of molecules its resident microbes had the capacity to produce. «For reasons we don’t understand, arid soils turned out to harbor microbes capable of producing a greater diversity of compounds,» Charlop-Powers says. For this preliminary survey, Brady called on his family to send in samples from Arizona and New Mexico; another postdoc in his lab, Jeremy Owen, collected soil in New England.
The Brady Lab would like to extend this study and hopes to encourage citizen scientists to contribute to the effort. The lab has set up a website: www.drugsfromdirt.org and after signing up, citizen scientists will receive information about how to collect and ship samples. The process is simple, says Brady: «Take a sandwich bag, a spoon or a trowel, and dump a couple of spoonfuls in the bag and ship it to us.»
Story Source: The above story is based on materials provided by Rockefeller University. Note: Materials may be edited for content and length.
Journal Reference: Z. Charlop-Powers, J. G. Owen, B. V. B. Reddy, M. A. Ternei, S. F. Brady. Chemical-biogeographic survey of secondary metabolism in soil. Proceedings of the National Academy of Sciences, 2014; 111 (10): 3757 DOI: 10.1073/pnas.1318021111
Cite This Page: MLA, APA, Chicago
Rockefeller University. «Searching for drugs in dirt: Researchers call on citizen scientists.» ScienceDaily. ScienceDaily, 16 May 2014. <www.sciencedaily.com/releases/2014/05/140516202659.htm>.
Related Articles: Oily fish, Bacteria, Biopharmaceutical, Drug discovery, Carbon monoxide. Kiwifruit
Chemical-biogeographic survey of secondary metabolism in soil
Abstract del Trabajo Original.
Significance
A comparative analysis of conserved domain fragments amplified from nonribosomal peptide and polyketide-type gene clusters present in diverse soil microbiomes revealed a link between soil type, species composition, and biosynthetic richness, as well as an unexpected conservation of the meta-secondary metabolome present within microbiomes from similar soil types. Although the functional consequences of these enrichment patterns is not yet clear, it suggests that similar soil types contain functionally related collections of secondary metabolites that likely play conserved roles in the ecology of these soils. This work provides a model for the large-scale extension of molecular phylogenetic-type analyses directly to the study, characterization, and comparison of secondary metabolite biosynthetic geneclustersthat remain hidden in diverse soil microbiomes.
Abstract
In this study, we compare biosynthetic gene richness and diversity of 96 soil microbiomes from diverse environments found throughout the southwestern and northeastern regions of the United States. The 454-pyroseqencing of nonribosomal peptide adenylation (AD) and polyketide ketosynthase (KS) domain fragments amplified from these microbiomes provide a means to evaluate the variation of secondary metabolite biosynthetic diversity in different soil environments. Through soil composition and AD- and KS-amplicon richness analysis, we identify soil types with elevated biosynthetic potential. In general, arid soils show the richest observed biosynthetic diversity, whereas brackish sediments and pine forest soils show the least. By mapping individual environmental amplicon sequences to sequences derived from functionally characterized biosynthetic gene clusters, we identified conserved soil type–specific secondary metabolome enrichment patterns despite significant sample-to-sample sequence variation. These data are used to create chemical biogeographic distribution maps for biomedically valuable families of natural products in the environment that should prove useful for directing the discovery of bioactive natural products in the future.

[…] suelo (directa o indirectamente) han producido o inspirado un gran número de fármacos que, como los antibióticos, durante decenios ha salvado millones y millones de vidas. La mayoría de estos bichitos no afectan a la salud humana, pero un pequeño grupo de ellos puede […]