![]()
Metabolismo del Suelo, Metabolismo Humano y Resistencia a los Antibióticos. Una Bioprospección Urgente
Ya os hemos comentado más de una vez que era un disparate científico no llevar a cabo una bioprospección del suelo y regolito, con vistas a conocer su biodiversidad y metabolismo. La realidad es tozuda, y a las pruebas nos remitimos. El post de hoy va a sorprender, tanto a los expertos como al ciudadano. Llevaba varios días esperando a hablar del tema, pero Salva González Carcedo se me ha adelantado, aunque afortunadamente no ha dicho todo.
- ¿Sabéis las enormes similitudes entre el metabolismo humano y el del suelo?
- ¿Sabéis que el metabolismo de este último es, con toda seguridad el más potente y diverso del planeta Tierra por unidad de área y volumen?
- ¿Sabéis que no hay antibiótico que se resista a las microbiomas edáficos?
Y ¿porqué es con toda seguridad el más biodiverso y potente por unidad de área y volumen?. La respuesta la encontraréis cuando, como nosotros, os preguntéis: ¿Cuanto mide un metro cuadrado de suelo? Si queréis un atajo leer el post enlazado. En cualquier caso, para los interesados en el tema, en este Pdf se explica la composición y estructura del microbioma intestinal. Impresionante. Ya os comentamos en otro post anterior como no podremos ganar la batalla a las bacterias con los antibióticos, por cuanto la selección natural nos lo impide, debido a la denominada guerra armamentística. Vayamos pues a las pruebas experimentales. Como podréis observar, el metabolismo de las comunidades microbianas deparará enormes sorpresas con importantes aplicaciones, cuando nuestros políticos científicos inviertan en lo que deben y no en lo que les genera más beneficios mediáticos. Si lo hacen, la ciencia del suelo pasara de ser hermana pobre a una estrella en el firmamento de las ciencias. Del mismo modo, resulta más que interesante aceptar que las comunidades microbianas que albergamos en nuestros intestinos atesoran un genoma más variado que el del propio ser humano, aspecto que ya aventuramos en nuestra entrega: individuos-ecosistemas.
La obesidad. Introducción. Una nueva perspectiva bacteriana.
El intestino humano es el habitat cuyo microbioma reúne un número 10 veces superior (entre 10 y 100 trillones) a las del conjunto de células somáticas y germinales que conforman nuestra organización de humana y su metagenoma es 100 veces mas rico que el nuestro. Steven Gill, del Institute for Genomic Research de la Universidad de Stanford, (2006) indica que en el interior del intestino, ya se han descrito unas 70 divisiones de Bacterias y 13 divisiones de Arqueas diferentes. Pero aparecen como dominantes dos filum bacterianos específicos: «Bacteriodetes» y “Firmicutes”, que incluyen cientos de filotipos.
En un reparto general, el 90% pertenecen a las dos filum indicados, y el 10% restante lo ocupaban las arqueas destacándose en especial, una metanogénica consumidora de H2 (Methanobrevibacter smithii), Euryarchaeota que supone hasta el 10% de todos los anaerobios presentes en el colon de un adulto sano.
E. Coli: una de las bacterias más frecuentes en
nuestros intestionos. Fuente: VentureBeat
Con la alimentación, llegan al intestino importantes aportes de fibra (polisacáridos), grasas y proteínas, que son degradados por exo-enzimas que están codificadas por los genes de los colectivos que conforman el microbioma intestinal (igualito que en el suelo) y de los que el genoma humano carece (Sonnenburg, et al. 2005; Gill, et al. 2006). Los productos de demolición estructural y degradación molecular generados por la actividad exoenzimática bacteriana, son su base nutricional en el intestino, lo que les permite el desarrollo de fermentaciones bacterianas endometabólico, y (…). En el hígado, estos nutrientes serán metabolizados, hasta adquirir conformaciones moleculares propias de organismo receptor (animal superior o humano).
Del mismo modo, el día 8 de Abril de 2008, el Boletín de noticias mi+d se hacía eco de una noticia firmada por Isabel Espiño en el “El Mundo Digital“: “Y la bacteria se comió al antibiótico”, que no tiene desperdicio con vistas a demostrar una vez más el impresionante metabolismo que el medio edáfico muestra. Sin duda alguna, podemos aseverar que es el más potente por unidad de área y volumen en el Planeta. Permítanme pues que reproduzcamos, prácticamente en su integridad, la mencionada noticia. Sin embargo, nunca debemos olvidar su fuente original (Science), que podéis encontrar en el siguiente enlace, aunque también conviene que leáis este otro comentario (que eso sí, esta escrito en Suahili): “Los Antibióticos como fuente de carbono para las bacterias” (texto parcialmente reproducido al final del post).
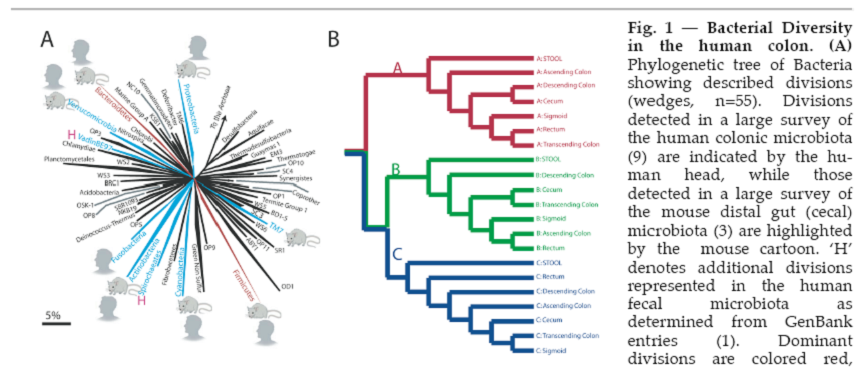
Diversidad del microbioma humano
Por paradójico que resulte, los antibióticos (esos fármacos diseñados para acabar con las bacterias) pueden ser un rico manjar para estos microorganismos. Un grupo de investigadores acaba de descubrir que multitud de bacterias del entorno son capaces de nutrirse de diferentes tipos de antibióticos. El hallazgo puede aportar nuevas claves sobre las resistencias a los antibióticos.
Se lo comen todo. «Prácticamente todas las moléculas orgánicas de la naturaleza pueden ser comidas por algún tipo de bacterias, por eso no vemos acumulaciones significativas de ningún tipo de material orgánico en el entorno«, explica a elmundo.es Gautam Dantas, uno de los autores del trabajo, que acaba de publicar la revista ‘Science‘.
Sin embargo, nadie se había planteado hasta ahora que en ese ‘todo’ también pudiesen encontrarse los antibióticos, sustancias sintéticas o naturales pensadas, precisamente, para matar bacterias. «Nuestro trabajo muestra que corren la misma suerte que cualquier otro compuesto orgánico del entorno», agrega este investigador, del departamento de Genética de la Harvard Medical School (EE.UU).
UNA AMPLIA VARIEDAD
Dantas y su equipo intuyeron este curioso fenómeno mientras realizaban una serie de experimentos sobre biocombustibles. Las bacterias están demostrando ser un útil sintetizador de estos carburantes y los investigadores recurrieron a los antibióticos durante experimentos que evaluaban el metabolismo bacteriano. «Nuestro nuevo estudio describe los resultados de un experimento más detallado que hicimos [a partir de aquel hallazgo] para evaluar estos patógenos en diferentes entornos y ante clases más variadas de antibióticos«, aclara el investigador.
En efecto, desde antibióticos clásicos como la penicilina a modernas moléculas como la ciprofloxacina resultaron ser una fuente de carbono (uno de los procesos metabólicos de las estos microoganismos) para multitud de bacterias del suelo. Los investigadores cultivaron cepas bacterianas de 11 orígenes diferentes en 18 antibióticos. Todos los fármacos facilitaron el crecimiento de algún tipo de bacteria y seis de ellos nutrían a bacterias de las 11 procedencias estudiadas. «Realmente, nos sorprendió la variedad de bacterias y, sobre todo, su ubicuidad y su capacidad para usar todos los antibióticos estudiados», aclara Dantas.
La más numerosa era la familia de proteobacterias Burkholderiales (41% de las 11 especies aisladas). En este grupo se encuentran también microorganismos que causan enfermedades en el hombre (como ‘Bordetella pertussis’, causante de la tosferina) y los animales (como ‘Burkholderia mallei’, culpable de una infección equina llamada muermo).
¿QUÉ PASA CON LAS BACTERIAS HUMANAS?
«Hemos demostrado que las bacterias que se comen antibióticos están ampliamente esparcidas por el entorno y constituyen un desconocido reservorio de genes [que regulan mecanismos responsables] de la resistencia a los antibióticos que pueden (o no) haber contribuido a los crecientes niveles de bacterias patogénicas resistentes a múltiples fármacos«, resume Dantas.
Es más, muchos de los patógenos que se nutrían de los antibióticos están estrechamente relacionados con patógenos que pueden afectar al hombre. «Dada la creciente incidencia de microbios patogénicos resistentes a múltiples fármacos, estábamos especialmente interesados en la relación entre estos microorganismos y las bacterias del entorno que sobreviven en antibióticos. Ahora, hemos continuado con algunos hallazgos excitantes sobre un fenómeno parecido en microbios humanos que planeamos difundir muy pronto», avanza el investigador (…).

Árbol Filogenético del metabioma microbiano (foto)
Seguidamente os mostramos el resumen original de la revista Science:
Bacteria Subsisting on Antibiotics
Gautam Dantas,1* Morten O. A. Sommer,1,2* Rantimi D. Oluwasegun,1 George M. Church1
Antibiotics are a crucial line of defense against bacterial infections. Nevertheless, several antibiotics are natural products of microorganisms that have as yet poorly appreciated ecological roles in the wider environment. We isolated hundreds of soil bacteria with the capacity to grow on antibiotics as a sole carbon source. Of 18 antibiotics tested, representing eight major classes of natural and synthetic origin, 13 to 17 supported the growth of clonal bacteria from each of 11 diverse soils. Bacteria subsisting on antibiotics are surprisingly phylogenetically diverse, and many are closely related to human pathogens. Furthermore, each antibiotic-consuming isolate was resistant to multiple antibiotics at clinically relevant concentrations. This phenomenon suggests that this unappreciated reservoir of antibiotic-resistance determinants can contribute to the increasing levels of multiple antibiotic resistance in pathogenic bacteria.

Los Antibióticos como fuente de carbono para las bacterias
(Geobacter sp.) (Fuente Betesize Bio)
Antibiotics as a Carbon Source
Here’s the context: “Eighty years after Alexander Fleming discovered penicillin on a moldy culture dish, the battle against killer bugs is faltering. More and more bacteria – including insidious tuberculosis strains that have cropped up2 – now shrug off almost all antibiotics. Meanwhile, few new antibiotics are reaching the clinic. Medicine is on the defensive, says microbiologist and physician Stuart Levy of Tufts University School of Medicine in Boston. ‘We are not keeping up with the bacteria.’” Antibiotic resistance has been a remarkable instance of natural selection in progress.
Now there is something even more remarkable concerning the seemingly endless capacity for microbes to do just about anything biochemically. They can even bacteria that can survive with nothing to eat but antibiotics. It’s not clear why the soil bacteria examined by Geneticist George Church and team appear particularly skilled at converting cytotoxic antibiotics into a carbon source, nor whether this is connected to the development of antibiotic resistance in human-specific pathogens – it’s just plain surprising.
But perhaps we shouldn’t be too surprised. We’ve known for a few years now that the primary microbial antibiotic-resistance mechanisms include efflux pumps, target gene-product modifications, and enzymatic inactivation of the antibiotic compound. We’ve also known for a few years that microbes can biochemically degrade just about anything, including cyclic hydrocarbons of probably any flavor, as well as bioremediation of heavy metals and radionuclides. [Incidentally, the image above is of Geobacter metallireducens as posted at Green Brooklyn]
While the biochemical wonders that microbes can spawn are amazing, and can be good for cleaning up our environment, what about the instance that began this post – microbial subsistence on antibiotics (much less resistance)? As I mentioned, this is a clear example of both the power of natural selection and the versatility of biochemistry. How then do we use our knowledge of these things to better control the pathogens that we seek to eradicate with antibiotics?
We have to use antibiotics more wisely, and perhaps almost not at all, just as we learned to get by without insecticides half a century ago in agriculture. Just as better ecology was the answer then, microbial ecology is an answer now. And an even better answer is to find ways to boost the best tool for combating disease that natural selection has given us: our immune system. (…).
Existe una canción popular española que hacía referencia, con mucho humor, a la guerra de que mantuvimos en este país a la hora de expulsar al ejército napoleónico, a principios del siglo XIX. Parafraseándolo podríamos decir que los microorganismos del suelo nos susurran burlones: “Con los antibióticos que nos tiran los fanfarrones, nos hacemos las bacterias tirabuzones” El día que lo aceptemos, emprenderemos un combate más certero contra la “resistencia” que nos presentan ese ejército de minúsculos e insignificantes (¿?) organismos. Hoy por hoy, entre ellas y nosotros, no puede haber ganadores ni perdedores, sino una mera escalada armamentística.

Juan José Ibáñez

es exelente saber que personas se interesan por la tierra, pero hay algo que me llamo la atención en el ser humano como reacciona ante un producto natural que si tiene resultados.
Como incrementa la respiración celular por medio de la reación química que proporciona el oxígeno a nivel celular.
nada perdemos con conocer tan exelente producto gracias.
creo que es muy buena la pagina trae todo lo q uno necesita en el colegio lo q le pidan
gracias pagina
aiozz!!
«Hoy por hoy, entre ellas y nosotros, no puede haber ganadores ni perdedores, sino una mera escalada armamentística.»
Sr. Juan José, tenga ud. la seguridad de que con este enfoque mal parados vamos a salir todos ( ya lo estamos). En vez de combatir a los microbios, a los que echamos la culpa de «casi todo» ¿no sería mejor intentar comprender la causa? ¿Cuáles son las razones por las que un microbio se vuelve un patógeno peligroso para la salud? Más teniendo en cuenta que forman parte de nosostros y son indispensables.
También resulta interesante saber que razones ¿sociológicas tal vez? motivan a los científicos a expresarse con una terminología militar. No recuerdo quien dijo eso de que la civilización comenzará el día en el que los humanos dejemos de darnos palos y comencemos a hablar. ¿No podríamos «hablar» con los microbios en vez de intentar exterminarlos?
Saludos
[…] Metabolismo del Suelo, Metabolismo Humano y Resistencia a Antibióticos – En caché […]
[…] Metabolismo del Suelo, Metabolismo Humano y Resistencia a los … […]