![]()
La Biodiversidad Del Suelo y su Subestimación por la Ciencia Contemporánea (Evidencias)
Ya os hemos comentado en varios de los post almacenados en nuestra categoría «Biología y Ecología del Suelo», que la biodiversidad que atesora el medio edáfico se encuentra enormemente subestimada por diversas razones. Si hablamos de la fauna, por ejemplo, os mostramos como cuatro métodos de extracción de nematodos ofrecen otras tantas imágenes francamente distintas de las estructuras de las nematocenosis. Del mismo modo, los microbiólogos reconocen desde hace décadas que muchas “especies” microbianas no se desarrollan en los cultivos de laboratorio. La nota de prensa y el artículo (revista PLoS One) que analizaremos hoy, dan cuenta de ello. Si es así, ¿Qué novedad ofrecen? Los Taxónomos en el ámbito de la microbiología llevan utilizando desde hace mucho tiempo la técnica conocida como PCR (análisis del ADN mitocondrial) con vistas a hacerse “una idea aproximada” de tal diversidad. Sin embargo, el estudio comentado demuestra la mentada subestimación (de ser corroborada), pero al mismo tiempo aporta algo más concreto y preciso. Las especies no detectadas resultan ser las menos abundantes en número o biomasa, es decir las que pertenecen al margen derecho de la Curva de Willis, ubicua a todos los inventarios de biodiversidad y edafodiversidad. Realmente, en cualquier inventario, son los taxa de este tramo de la curva los que, con toda seguridad, sufren más riesgos de pasar inadvertidos. Lo raro y escaso es siempre más difícil de detectar. Se trata de una obviedad. Ahora bien la investigación de marras parece abrir una ventana con vistas a desarrollar tecnologías más precisas en el futuro. Mientras tanto, al menos será viable cuantificar hasta que grado los estudios con PCR subvaloran la biodiversidad “taxonómica real” de un suelo. También os hemos comentado que existen instrumentos tecnológicos en el mercado que aseveran que puede cuantificarse el número de especies microbianas con bastante precisión, como es el caso del PhyloChip, del que os hemos hablado en dos post precedentes (puro marketing) y otro más original que precedrá a este: «el sistema inmune de las plantas se encuentra en los suelos«.
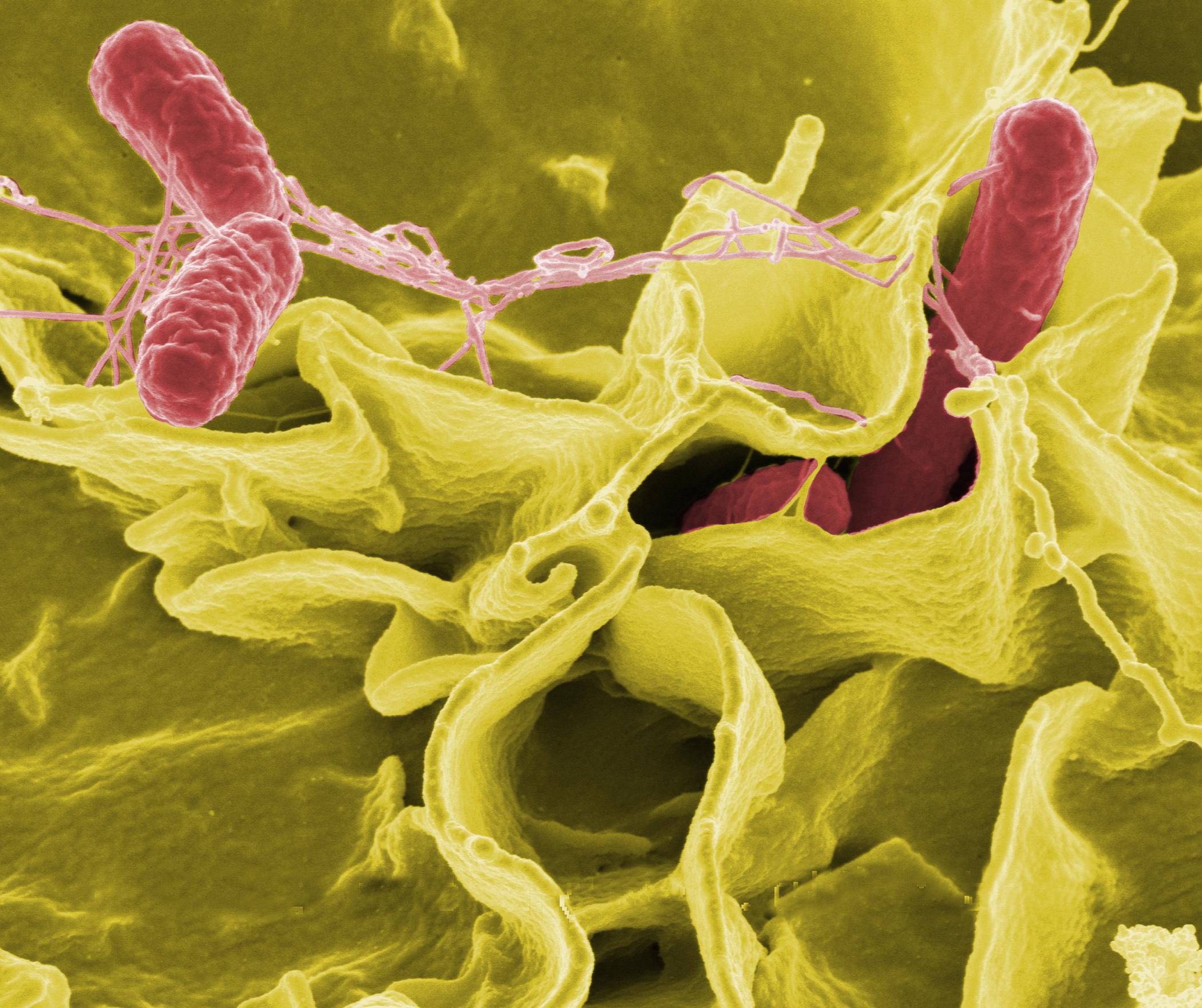
Comunidades Microbianas del Suelo. Fuente: Computacional Reserach, Berkeley Lab
Francamente me alegra hablar de estudios realizados por españoles pertenecientes a mi institución, y más aun cuando pertenecen a los antiguos centros de edafología y Agrobiología, en los cuales se están desmantelando la ciencia del suelo por decisión de nuestras autoridades. Pues bien, para finalizar y dar paso a la nota de prensa y el abstract de la publicación digamos, que no se trata tan solo de un análisis que concierna a la ecología del suelo sino que parece ocurrir lo mismo, para la plétora bichitos que atesoramos en nuestro cuerpo. Hablamos del denominado microbioma humano. En otras palabras, existen muchos microorganismos en nuestro cuerpo que aun desconocemos, y como corolario tampoco las repercusiones que tienen en nuestra salud-enfermedad.
Os recordamos que PLoS One es una revista en acceso abierto, por lo que podéis bajaros el artículo original sin coste alguno.
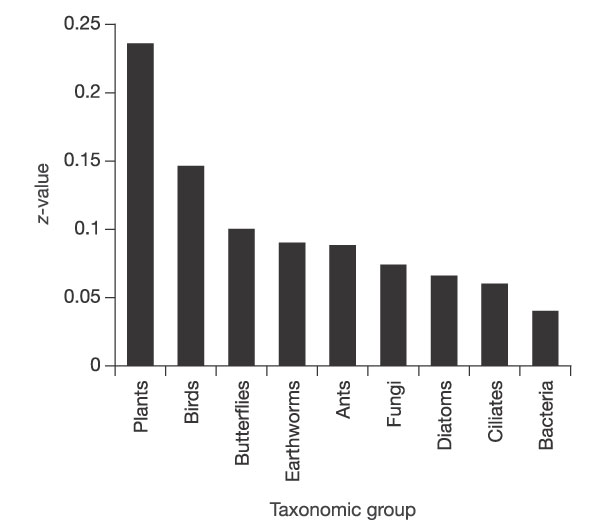
Curva de Willis de un Inventario, ya sea de biodiversidad o edafodiversidad. Fuente: Juan José Ibáñez
Juan José Ibáñez
La técnica estrella para detectar bacterias discrimina las más raras Mejor que el PCR
Un equipo con participación del Consejo Superior de Investigaciones Científicas (CSIC) ha comprobado que la amplificación por la reacción en cadena de la polimerasa o PCR (por sus siglas en inglés), el método más empleado actualmente para la detección de bacterias, discrimina las menos abundantes.
FUENTE | CSIC – mi+d; 16/01/2012
El estudio, que aparecerá publicado en el próximo número de PLoS One, confirma que no es posible determinar la estructura de la enorme diversidad microbiana con los métodos actuales.
Rubén Duro Pérez
La reacción en cadena de la polimerasa o PCR se emplea como técnica molecular para amplificar o generar múltiples copias de los genes de ARN ribosomal de un microorganismo específico. “En este estudio hemos comprobado que la detección de bacterias es dependiente de la fracción que representan respecto al total de la comunidad microbiana”, señala Juan Miguel González, investigador del CSIC en el Instituto de Recursos Naturales y Agrobiología ubicado en Sevilla.
Los científicos, en colaboración con un equipo del Centro Superior de Investigación en Salud Pública de Valencia, han empleado para los experimentos sondas fluorescentes específicas y dos cebadores (una cadena de ácido nucleico como punto de partida para la replicación de ADN). Tras amplificar cada tipo de bacteria simultáneamente en la misma reacción, observaron que la eficiencia de amplificación para las abundantes era elevada, mientras que para las que representaban una fracción reducida era muy baja.
CONOCER LOS RIESGOS
“La diversidad microbiana, que es la base que sustenta la búsqueda de nuevos biocatalizadores y recursos para la explotación biotecnológica, es probablemente muy superior a lo que somos capaces de detectar actualmente. Conocer el riesgo de los sesgos introducidos en la detección y cuantificación de secuencias amplificadas es algo importante mientras se continúe utilizando la amplificación por PCR o hasta que pasemos a una tercera generación de métodos de secuenciación”, señala González.
Referencia Bibliográfica
Juan M. González, Maria C. Portillo, Pedro Belda‐Ferre, Alex Mira. Amplification by PCR artificially reduces the proportion of the rare biosphere in microbial communities. PLoS One DOI: 10.1371/journal.pone.0029973
Juan M. Gonzalez, Maria C. Portillo, Pedro Belda-Ferre2,Alex Mira
Institute of Natural Resources and Agrobiology, Spanish Council for Research, IRNAS-CSIC, Sevilla, Spain, 2 Genomics and Health Department, Center for Advanced Research in Public Health (CSISP), Valencia, Spain
Enlace para leer el artúculo completo y bajarlo in costo alguno de Internet
http://www.plosone.org/article/info%3Adoi%2F10.1371%2Fjournal.pone.0029973
Abstract: The microbial world has been shown to hold an unimaginable diversity. The use of rRNA genes and PCR amplification to assess microbial community structure and diversity present biases that need to be analyzed in order to understand the risks involved in those estimates. Herein, we show that PCR amplification of specific sequence targets within a community depends on the fractions that those sequences represent to the total DNA template. Using quantitative, real-time, multiplex PCR and specific Taqman probes, the amplification of 16S rRNA genes from four bacterial species within a laboratory community were monitored. Results indicate that the relative amplification efficiency for each bacterial species is a nonlinear function of the fraction that each of those taxa represent within a community or multispecies DNA template. Consequently, the low-proportion taxa in a community are under-represented during PCR-based surveys and a large number of sequences might need to be processed to detect some of the bacterial taxa within the ‘rare biosphere’. The structure of microbial communities from PCR-based surveys is clearly biased against low abundant taxa which are required to decipher the complete extent of microbial diversity in nature.
Citation: Gonzalez JM, Portillo MC, Belda-Ferre P, Mira A (2012) Amplification by PCR Artificially Reduces the Proportion of the Rare Biosphere in Microbial Communities. PLoS ONE 7(1): e29973.

[…] realizar comparaciones rigurosas. Nuestro último post sobre este tema llevaba por título: “La Biodiversidad Del Suelo y su Subestimación por la Ciencia Contemporánea”, aunque hemos escrito muchos más, como este otro que data de hace seis años: “El […]
[…] inherentes con vistas a estimar la biodiversidad del Suelo, por lo que esta se encuentra muy subestimada. Del mismo modo, mentamos que ciertos tipos de organismos biológicos son tan abundantes y ubicuos […]